Spectrometrie mase Maldi-TOF. Origins
Introducerea desorbției cu laser asistată de matrice (MAS) a fost, în orice caz, schimbarea tehnologică a celui mai mare proiect a avut loc în clinica de microbiologie din ultimul deceniu. În câțiva ani a trecut de la a fi o noutate promițătoare, a fi o tehnologie complet integrată în activitatea clinică zilnică și disponibilă, în țara noastră, în serviciile de microbiologie ale numeroaselor centre spitalicești1. Pentru aceasta, a fost necesar ca spectrometrele de masă să sufere o evoluție tehnologică semnificativă. În acest sens, au existat două progrese cruciale. În primul rând, proiectarea timpului sistemului de zbor (TOF) de către W.E. Stephens, în 1946, care a permis separarea diferitelor mase. Atunci când accelerează ionii într-un câmp electric și dobândește toată aceeași energie cinetică, viteza pe care o dobândesc și, prin urmare, timpul folosit pentru a călători tubul de vid, depinde de masa moleculei ionizate, care poate fi dedusă din timpul petrecut Această rută.
A doua piatră de hotar a fost dezvoltarea metodelor care au permis proteine intacte ionizante care, datorită dimensiunii lor, până atunci nu au fost susceptibile la ionizare, deoarece energia ridicată necesară pentru el a ajuns să modifice modificarea sau distrugând propria proteină. În 1987, Köichi Tanaka prezintă o nouă metodă de analiză (desorbție laser moale) care a permis transferarea la molecule energia necesară pentru a le ioniza fără a rupe legăturile chimice fragile. Această descoperire ia câștigat Premiul Nobel în chimie în 2002, împreună cu John B. Fenn, pentru „dezvoltarea metodelor de identificare și analiză structurală a macromoleculelor biologice”. Nu mai existau spectrometre de masă pe baza acestui tip de analiză, deși au fost necesare unele schimbări în metodă, astfel încât să apară în cele din urmă Maldi-TOF, în care desorbția ionilor este favorizată de o matrice care absoarbe energia laserului și îl transferă parțial la obiectul de studiu al moleculelor. Chiar dacă aceste modificări au îmbunătățit tehnica, făcându-l mai ușor și mai ușor, cei responsabili pentru aceste îmbunătățiri, Michael Karas și Franz Hillenkamp, nu au fost incluse în Premiul Nobel, care a generat o controversă importantă. Pe baza contribuțiilor acestor autori au început să fie posibilă, studiul macromoleculelor și biopolimerii cu MS, care a deschis noi domenii de aplicare pentru această tehnologie.
în aproape 30 de ani de viață, Maldi em – TOF a fost utilizat pentru analiza cantitativă și calitativă a proteinelor de diferite origini. Inițial, a fost aplicată proteine izolate anterior sau la seturi mici de proteine, dar datorită progreselor tehnice atât în instrumente, cât și în instrumentele pentru analiza computerizată a datelor, poate fi acum abordat studiul grupurilor mari de proteine. /p>
Posibilitatea de a începe să studieze proteine complexe, și nu doar peptide mici, au extins foarte mult câmpurile potențiale de aplicare a MS. Unul dintre ei a fost identificarea microorganismelor că, deoarece procedura a fost simplificată și software-ul necesar a fost îmbunătățit pentru a exploata datele brute furnizate de spectrometru, derivate rapid în aplicația clinică
până la introducerea EM Maldi-TOF, identificarea bacteriană, chiar și cu progrese semnificative, cum ar fi crearea galeriilor de identificare miniaturizate și automatizarea inoculării și a lecturii, au continuat să fie hrănite prin metodele dezvoltate de bacteriologia clasică Practica sistemelor de identificare a continuat să se bazeze pe fermentarea zaharurilor și pe detectarea acestora prin schimbarea pH-ului generat, metabolizarea altor substraturi și producția de metaboliți diferiți și activități enzimatice detectabile prin metode chimice. Toate aceste metode au adus mai multe limitări:
- •
Necesită creșterea bacteriană, care se presupune, în majoritatea cazurilor, o perioadă de incubație de cel puțin 16-18 ore din inocularea sa până la citire. /p>
- •
a arătat probleme de identificare în toate acele microorganisme cu dificultăți de creștere în mediile lichide utilizate pentru inocularea acestor panouri, precum și în microorganisme cu activitate biochimică și enzimatică scăzută. fost necesar să se ia în considerare marja de eroare derivată din faptul că, indivizii aceleași specii pot avea comportamente diferite împotriva diferitelor substraturi.
Aceste limitări, deși au fost cunoscute și asumate, ele au devenit mai clar brevete când, având în vedere discrepanțele observate în unele studii între ID-ul MSZ-TOF și identificarea convențională, Secvențierea RNR 16 au arătat că, în marea majoritate a cazurilor, identificarea corectă corespunde celor furnizate de emalia EM MALDI-TOF2.
în acest sens, EM Maldi-TOF are avantaje evidente:
- • •
-
Analiza profilului proteic al microorganismului în spectrul 2- 20kd, care este locul în care sunt plasate majoritatea proteinelor de nervuri Osomic, oferă pentru marea majoritate a speciilor bacteriene un profil specific, care permite diferențierea de la restul cu o fiabilitate similară celei oferite de secvențierea RNR 16S.
Indiferent de posibilitatea de a identifica microorganismele direct de la unele eșantioane, care este tratată într-o altă secțiune, chiar și o creștere foarte scăzută sau precoce, permite o identificare fiabilă într-o perioadă scurtă de timp , economisind astfel cel puțin acele 16-18 ore de creștere a sistemelor de identificare biochimice.
A Acest lucru trebuie adăugat că introducerea acestei tehnologii, a extins considerabil gama de genuri și specii pe care le putem identifica cu fiabilitate, cu o metodă capabilă să fie utilizată în rutină. Acest lucru a dus chiar la o reevaluare a rolului ca agenți patogeni de microorganisme care, datorită dificultății identificării lor prin metodele clasice, au fost cel mai probabil infradiagnosed3.4.
deja în 1975, Anhalt și Fenselau propune utilizarea MS pentru identificarea microorganisme5. Douăzeci de ani mai târziu, primul studiu este publicat care demonstrează eficacitatea Maldi-TOF EMS pentru identificarea microorganismelor din celulele complete6.
În 2009 a fost publicată, probabil, un articol cheie pentru a da cunoașterea în general , specialiști implicați în diagnosticarea bolilor infecțioase, posibilitățile EM MALDI-TOF7. Studează mai mult de 1600 de izolate care includ bacterii de agitare și gramnegative, aerobic și anaerob, obținând 95,4% din identificări corecte. Acest articol vizează acest articol care a fost transcendent pentru utilitatea practică a acestei metodologii: disponibilitatea bazelor de date cu microorganisme suficient de largi, atât din punct de vedere calitativ (numărul de genuri, cât și din speciile incluse) și cantitative (demonstrează autorii că probabilitatea corectă de identificare este mai mare pentru acele microorganisme care există cel puțin zece profiluri diferite introduse în baza de date).
Din acest moment, expansiunea rapidă a utilizării are loc de această tehnologie în microbiologia clinică. Prima publicație din Spania are loc în 2010, arătând o corelație cu metodologia convențională, la nivelul speciilor, 100% în gramatici și 87,7% în gramnegativ8. De atunci, numărul de publicații referitoare la diferite aspecte ale utilizării clinice a emaldi-TOF, dar mai presus de toate la identificarea microorganismelor, a fost exponențială7-12.
Identificarea bacteriilor gramnegative utilizând Maldi-TOF
Studiile arată că eficiența în identificarea enterobacteriei și a altor gram-negative este excelentă, inclusiv cele mai frecvente gram negative în clinic, cât și cele mai puțin frecvente sau mai complicate pentru a identifica cu metodologia clasică (diferite specii de yersinia, aeromonas, plasiomonas ,. Brucella, Francisella, Achromobacter, Stenotirofomonas, Burkholderia …). Ca și în alte cazuri, problemele de identificare au fost aproape întotdeauna legate de insuficiențele bazelor de date, mai degrabă decât limitările metodei, așa cum sa întâmplat în unele studii cu genuri cum ar fi Ralstonia, Elizabethkingia sau Sphyngobacterium13. Inițial, au fost ridicate unele limitări care ar putea avea o mai mare transcendență clinică, cum ar fi incapacitatea de a face diferența între Escherichia coli și Shigella14 sau dificultatea de a diferenția serovarii de Salmonella Entertica. Studiile mai recente sugerează că este posibil să se salveze aceste limitări. Folosind programe de calculator, cum ar fi flexanaliză și clinkools (Bruker Daltonics GmbH, Germania), au fost identificate vârfuri specifice care să permită diferite de la E. coli și Shigella în 90% din cazuri15. Alți autori au arătat că această diferențiere este posibilă chiar și fără a utiliza niciun software suplimentar, pur și simplu creșterea numărului și a varietății profilurilor E. coli și Shigella prezente în baza de date de referință16. Cu această metodă, autorii identifică corect 60/64 izolate de E. coli și izolate de 110/116 ale lui Shigella.
Pentru toți cei care au folosit EM Maldi-TOF pentru identificare, se constate că, cu metodologia obișnuită, identificarea Salmonella la un nivel de gen este fiabilă, dar dincolo de nivelul de gen este mult Mai puțin. Cu toate acestea, există deja din 2004 publicații care sugerează existența unor vârfuri specifice care ar putea permite identificarea serovarilor cu o mai mare fiabilitate17. Un studiu recent sugerează existența vârfurilor care permit identificarea în mod fiabil Typhi18 Serovar. Deoarece elementele diferențiale par să fie existente, o prelungire a bazei de date de referință cu un număr mai mare de difuzoare, cel puțin serovarii cele mai frecvente, ar îmbunătăți probabil rezoluția în acest caz particular.
în alte cazuri, La fel ca și diferențierea dintre speciile complexului Enterobacter Cloacaee, capacitatea EM Maldi-TOF este de aproximativ 80%, care, fără a fi optimă, reprezintă o îmbunătățire sensibilă față de metodologia convențională19.
pe Altfel, după cum sa menționat deja, identificarea corectă a microorganismelor identificate incorect, cum ar fi genul Raultella, identificate frecvent prin metode clasice, cum ar fi Klebsiella, fie ca enterobacter, permite o viziune mai reală a rolului său de agenți patogeni, subevaluate anterior3.
Identificarea bacteriilor de grampă utilizând MALDI-TOF
Identificarea microorganismelor gram-pozitive arată, de obicei, figuri oarecum mai mici, datorate În unele cazuri, la dificultatea de a culca zidul și în altele la similitudinea dintre specii. În general, metoda directă (aplicarea coloniei împreună cu matricea de pe placa de spectrometru) este de obicei suficientă pentru a obține o bună identificare din colonie, dar uneori extracția anterioară cu etanol / acid formic poate fi necesară pentru a obține valori optime de identificare. Împreună, oferă rezultate excelente în identificarea Staphylococcus aureus și a altor specii și a subspecii Staphylococi, atât producători, cât și de non-producători de coagulasă, precum și de la enterococi și alte decese mai puțin frecvente ca micrococus, gemella, Rothia etc. De asemenea, și-a arătat utilitatea în identificarea baciliului gram pozitiv, incluzând o identificare complexă prin metode clasice (Listeria, Lactobacillus, Corynebacterium, Nocardia, Actinotignum …) 20.
în plus, ca în Cazul gramnegativelor, utilizarea sistematică a acestei tehnologii se schimbă ca agenți patogeni ai unor microorganisme, cum ar fi Staphylococcus lugdurensis21. Împreună, datele disponibile fac recomandabil, în cazul stafilococi de utilizat, ca în orice alt caz, bazele de date care oferă un număr mai mare de specii și un număr rezonabil de profile de către specii și să utilizeze o metodă de extracție în loc de Aplicație directă, precum și aplicarea microorganismului pe placă în duplicat. Reducerea scorului necesar pentru validarea identificării la nivel de specii la valori > 1.7, în loc de > 2.0, deoarece este obișnuit , deoarece unii autori22 propune, poate crește numărul de identificări la nivelul speciilor, dar în schimbul unei scăderi a specificității a cărui relevanță poate fi discutabilă.
Probabil grupul de streptococci este cel care prezintă probleme mai mari. între gramici. În timp ce unele dintre speciile patogene principale (Streptococcus Pyogenes, Streptococcus agalactiae) sunt identificate cu fiabilitate ridicată, streptococcus pneumoniae (S. pneumoniae) a fost întotdeauna o sursă de probleme, datorită confuziei sale cu alte specii din grupul Mitis. Principalele echipe utilizate în țara noastră identifică S. pneumoniae cu fiabilitate. Cea mai obișnuită eroare este identificarea eronată a altor streptococi a grupului Mitis, cum ar fi S. pneumoniae, în special cu baza de date Bruker23. Unele vârfuri au fost descrise ca S. pneumoniae, astfel încât unii autori recomandă utilizarea lor pentru a corobora aceste identificări23. Cu toate acestea, ultima bibliotecă de referință a Bruker Daltonics (biblioteca MBT 6903 MSP) continuă să recomande utilizarea altor teste (Optochina, solubilitatea în bilă) pentru a le diferenția.
chiar și în același grup, calitatea de calitate a calității Identificarea diferitelor specii poate varia considerabil, așa cum se întâmplă în cadrul grupului Streptococcus Anginosus.
în orice caz, ca și în cazul Staphylococci, utilizarea unei baze de date largi și complete posibil și identificarea în duplicat și cu protocoalele care includ extracția sunt recomandate pentru a optimiza rezultatele (fig.1).
Adabitiuni, gram-negative, anaerobi și prelucrarea drojdielor utilizând Maldi-Tof.
Identificarea bacteriilor Anaerobias prin Maldi-Tof
Identificarea bacteriilor anaerobe, atât gramos, cât și gramnegative, are un grad ridicat de fiabilitate, identificând corect cele mai comune specii (bacteroide, precipotella, porfiromonas, fusobacterium, Clostridium, Actinomyces …). Aplicarea EM MALDI-TOF în acest domeniu a arătat, de asemenea, că identificarea biochimică a fost mult mai puțin fiabilă decât a fost gândită3. Combinația de simplitate, rapiditate și fiabilitate face ca EM Maldi-TOF în metoda de alegere pentru identificarea de rutină a bacteriilor anaerobe în clinică. Extracția sau nu eșantionul nu pare să modifice semnificativ rezultatele, nici nu face mediul de cultură sursă. Un factor decisiv, ca și în alte cazuri, este amploarea bazelor de date. Deja unul dintre primele studii efectuate în Spania3 a arătat că majoritatea erorilor în identificarea bacteriilor anaerobe au fost datorate absenței profilurilor de referință ale speciilor corespunzătoare din baza de date. Studiile mai vechi oferă o eficacitate în identificarea a 75-80%, în timp ce studii mai recente, cu baze de date complete sau baze de date specifice oferă cifre de peste 90%. Abilitatea de a identifica ribotipurile Clostridium difficile, sugerată de unii autori, nu a fost demonstrată definitiv.
Identificarea drojdiei și a ciupercilor filamentous prin Maldi-TOF
Identificarea drojdie a fost prezentată de la început rezultate excelente, diferențiate chiar și Specii complicate de discriminare prin metode convenționale, cum ar fi complexul de parapsihoză Candida. Cu toate acestea, rezultatele în legătură cu ciupercile filamentoase au fost mult mai neregulate. Unele studii inițiale au arătat rezultate bune care studiază selectiv sporii, dar această metodă este impracticabilă într-un laborator clinic, astfel încât majoritatea studiilor au fost direcționate către studiul comun al sporii și hife. În cazul ciupercilor, extracția convențională cu acid formic și acetonitril îmbunătățește semnificativ rezultatele. Alte metode care au fost testate, cum ar fi utilizarea margelelor de sticlă la fragmentul pereților, nu par să ofere rezultate semnificativ superioare extracției convenționale.
O problemă pusă de ciuperci filamentous este existența unor diferențe semnificative În profilurile proteice obținute în conformitate cu antichitatea culturilor și chiar între subcultura diferită a aceluiași CEPA24. Soluția la acest lucru trece prin elaborarea bazelor de date mai largi și mai complexe, care încorporează profiluri dintr-un număr mai mare de tulpini și culturi de antichitate diferită. Unii producători recomandă eliminarea după o recoltă de o zi în bulion, deși acest lucru are un impact asupra unei întârzieri de 24 de ore în emiterea rezultatului. Un studiu realizat în 201125 arată că o bază de date bine elaborată și suficient de complexă este fundamentală și că poate îmbunătăți identificarea ciupercilor filamente la cifrele similare cu cele obținute cu bacterii și drojdie. Din păcate, elaborarea și validarea acestor baze de date sunt complexe și nu sunt disponibile pentru mulți utilizatori, iar puținele dezvoltate sunt disponibile. Prin urmare, și, de asemenea, în scopuri de standardizare, este probabil mai adecvat să se utilizeze bazele de date ale producătorilor, care trebuie extinse convenabil.
În prezent Bruker Daltonics, a cărui bază de fungi filamentous, în versiunea sa MBT 6903 MSP Biblioteca , include 25 de specii Genera și 42, recomandă cultura în mediu lichid peste noapte, urmată de centrifugare, spălare și extracție cu etanol, acid formic și acetonitril (fig.2). MS-Vitek, în versiunea 3.0, include 32 de specii Genera și 81, iar Saramis în versiunea RUO 4.13 include o bază de date mai largă, cu 45 de genuri și 168 de specii. Biomérieux nu recomandă cultura în mediu lichid, ci o extracție convențională cu etanol, acid formic și acetonitril (figura 2). În ciuda tuturor, un studiu recent care utilizează metodologia recomandată de Bruker identifică corect, la nivelul speciilor, doar 72% din izolarea26 și un studiu foarte recent cu Vitek 3.0 identifică corect 66,8% din 318, datorită deficiențelor din baza de date27. Prin urmare, astăzi, identificarea fungiilor filamentous de către em Maldi-TOF nu poate înlocui complet metodologia de identificare convențională.
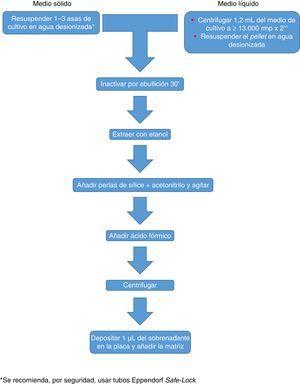
Prelucrarea mycobacteriană recomandată pentru sistemul Biotyper Maldi.
div id = „4895790F6A”
Identificarea micobacteriilor utilizând Maldi-TOF
Limitările metodologiei tradiționale de identificare a micobacterii, pe tot ceea ce se referă la timpul de răspuns, ei au făcut ca integrarea totalității laboratoarelor să fi decantate de metodele de identificare moleculară. Utilizarea EM MALDI-TOF pentru identificarea micobacteriilor este o alternativă la fel de rapidă și mai ieftină decât tehnicile moleculare, dar prezintă particularități în mai multe aspecte. Pe de o parte, metodele directe de analiză sau care implică o concentrație ridicată sau o manipulare a microorganismelor anterior inactivării sale nu sunt recomandate de o chestiune de siguranță biologică. Utilizarea unei metode de extracție care garantează ruperea celulelor îmbunătățește calitatea spectrelor și crește siguranța procedurii.
Mai mulți factori pot influența eficacitatea identificării micobacterii de către em Maldi – Tof. Așa cum sa arătat și cu ciuperci, Mycobacteria poate avea profiluri de proteine foarte diferite în funcție de antichitatea culturilor, deci este important ca diagnosticul să includă profiluri de proteine ale culturilor de antichități diferite pentru fiecare microorganism. De asemenea, sa vorbit despre posibila formare a polimerilor care maschează dimensiunea reală a proteinelor caracteristice utilizate pentru identificare. Toate acestea fac ca identificarea micobacteriilor să fie o procedură mai puțin previzibilă în ceea ce privește rezultatele sale decât cea a altor bacterii, după cum demonstrează eterogenitatea datelor publicate. În cele mai recente actualizări, bibliotecile au prezentat îmbunătățiri semnificative. Astfel, de exemplu, biblioteca micobacteriană a biotijului 3.0 include 149 de specii, iar versiunea 3.0 a MS Vitek încorporează 48 de specii noi în ceea ce privește versiunea anterioară.
Procedura recomandată inițial de Bruker a inclus o serie de etape (ajustarea la O anumită densitate optică, mai multe etape de centrifugare și resuspendare) care au presupus un risc biologic probabil inutil, astfel încât acestea au fost înlocuite cu o singură etapă de tratament cu căldură. Metoda recomandată include în prezent mai multe etape de încălzire și spălare, urmate de un tratament cu mingi de silice și acetonitril. Ulterior, ele sunt tratate cu acid formic, aplicate pe placă și acoperite cu matricea (figura 3). Dimpotrivă, metoda susținută de biomérieux parte a unui tratament inițial cu etanol de 70% și bile de silice, după care proba este extrasă cu acid formic și acetonitril, aplicată pe placă și acoperită cu matricea (figura 4).
Prelucrarea mycobacteriană recomandată pentru sistemul Vitek MS.
(0.15MB).
Procesarea fungică recomandată pentru Biotiper Maldi și sistemele Vitek MS.
NTE28 arată că, deși identificarea tinde să fie ceva mai bun pornind de la o jumătate de 7 ore decât Lowenstein Jensen, diferențele sunt mici în ambele sisteme. Cu toate acestea, aceasta demonstrează că metoda de procesare a eșantionului este critică și nu este interschimbabilă între sisteme. Probele obținute cu metoda de extracție a lui Bruper oferă rezultate mai bune în spectrometrul lor, iar prelucrarea cu metoda Vitek face acest lucru cu Salamis și MS Vitek în sine, deși diferența în acest caz este mai mică. Împreună, Vitek MS pare să fie mai puțin afectată de metoda de extracție. În ceea ce privește eficiența în identificare, Bruker Biotyper a identificat un scor ≥1,8 81,5% din izolate, în timp ce Saramis și Vitek MS 3.0 identificate cu o fiabilitate ≥90% cu 85,4 și respectiv 89,2%. Folosind aceleași criterii, 22,6% din izolatele identificate de Bruker Biotiper au trebuit să fie extrase pe mai mult de o ocazie pentru a obține identificarea, care au avut loc cu 21,5% cu Saramis și cu 14,2% cu VITEK MS 3.0.
În legătură cu alte microorganisme cu structură parietală apropiată de cea a micobacteriilor, pentru timp s-a considerat că identificarea sa a fost problematică datorită compoziției peretelui său. Cu toate acestea, s-a arătat că, dacă o bază de date este disponibilă cu suficiente referințe, o metodă de extracție similară cu cea utilizată cu Mycobacteria poate oferi rezultate bune29.
Toate datele referitoare mai sus sugerează că spectrul de microorganisme susceptibile la identificarea de către EM Maldi-TOF este extraordinar de largă și că, cu excepții, factorul de limitare a capacității emusurilor Maldi-TOF aproape întotdeauna mai mult în completitudinea bazelor de date de referință decât în capacitatea metodei de obținere a profilurilor fiabile de aproape orice microorganism bacterian capabil să crească în mediile de cultură. De fapt, companiile de producție par a fi conștiente de această problemă; Astfel, ultima bibliotecă de referință publicată de Bruker Daltonics (biblioteca MBT 6903 MSP) încorporează 938 de noi profiluri, dintre care 17,2% sunt destinate să acopere noi genuri și specii (15 genuri noi și 96 de specii noi), în special anaerobi și ciuperci, Dar 82,8% vizează creșterea diversității profilurilor din speciile deja avute în vedere în bibliotecile anterioare. În același mod, Vitek MS Versiunea 3.0 include 242 de specii bacteriene noi, printre care 48 de mici micobacterii și 15 nopți și 55 de ciuperci noi, cu o medie globală de mai mult de 10 izolate și 25 de spectre de specie.
Identificare de microorganisme, de Maldi-TOF, de la eșantionul direct
posibilitatea de identificare a microorganismelor înainte de creșterea colonii a fost, de la început, un obiectiv foarte atractiv, deci presupune momentul de a putea stabili a Tratament empiric mai orientat, reducerea apariției rezistenței și optimizarea cheltuielilor30. EM Maldi TOF, aplicat la eșantionul direct, a devenit un instrument foarte util în infecții severe, cu rată mare de morbiditate, cum ar fi bacteremia și fungemias, deși lucrările directe pe eșantioane au inconveniente care pot limita utilitatea tehnicii. În eșantioane cu densitate microbiană scăzută, nu este posibilă efectuarea unei identificări corecte, deoarece cantitatea de proteine bacteriene prezente este insuficientă. Se produce ceva similar cu probele polimetice, deoarece poate fi generat un spectru de proteină aberant, ca urmare a amestecului de mai multe profiluri sau ignorând direct microorganismul care se află într-o proporție minoră. Unele îmbunătățiri software au fost dezvoltate, care uneori pot detecta și chiar identifica microorganismele implicate separat, dar necesită a fi studiate cu atenție și, probabil, rafinate pentru a avea utilitate reală. În majoritatea metodelor care lucrează direct pe eșantion și înainte de posibila prezență a microorganismelor intracelulare, este recomandabil să se utilizeze metode care includ liza celulară.
Nu toate probele sunt valabile pentru acest tip de studiu. Eșantionul ideal este unul care provine dintr-o zonă sterilă obișnuită, care poate găzdui concentrații ridicate de microorganism și în care nu există limitări semnificative cu privire la volumul eșantionului. Probele din zonele colonizate în mod obișnuit (pielea, scaunul, aparatul de respirație ridicată …) vor genera profiluri aberante în toate probabilitățile. O problemă tehnică care trebuie luată în considerare atunci când lucrează direct pe eșantion este volumul de eșantion disponibil, care, în cazurile de urină sau culturile de sânge, nu este de obicei o problemă, dar în altele cu lichid cefalorahidian poate fi și că va fi la fel de Mult mai important ca minorul este concentrația bacteriană. În plus, eșantioanele cu conținut ridicat de proteine din surse străine la microorganism pot genera, de asemenea, probleme în timpul interpretării sale31.
Cu toate acestea, avantajul care este, în special în hemoculturile, depășește 24 de ore pe identificarea Microorganismul și orientarea tratamentului mai specific, a făcut ca utilizarea directă a MS pe eșantion, în special în aceste cazuri, a răspândit.
Identificarea directă din probele de urină
Urina este una dintre probele care se adaptează cel mai bine la Condiții ideale de a lucra cu EM Maldi-TOF pe eșantionul direct. Este un fluid steril în condiții fiziologice, încărcătura bacteriană în urina infectată este ridicată în majoritatea cazurilor, iar limitările privind volumul eșantionului și obținerea de probe noi, dacă este necesar, sunt minime.
Studii cu excelent Rezultatele au fost publicate, cu concordanță în identificarea care atinge 90-95% 32,33 comparativ cu sistemele automate.Diferitele protocoale de prelucrare a probelor au fost descrise, toate coincide în necesitatea unei pre-screening (citometrie de flux, colorarea gramului) pentru a discrimina între eșantioanele pozitive și negative și limitează utilizarea MS la eșantioanele inițial pozitive34.
Cu variații mici, prepararea probelor constă, de obicei, că le-a prezentat într-o serie de centrifugații și se spală cu apă deionizată, apoi efectuând procedura convențională a Maldi-TOF. Modificările au fost realizate prin încorporarea alicotului de probă SDS la 10% 33 pentru a îmbunătăți eliberarea proteinelor sau a Tween-8034 pentru a îmbunătăți rezultatele. Rezultatele obținute sunt, de obicei, mai bune cu gramnegative decât cu gramatici și ciuperci, îmbunătățind mult fiabilitatea atunci când numărul sunt ridicate, peste 105fc / ml.
Un inconvenient al acestei metode a fost nevoia de a efectua Studiu de sensibilitate prin metodologia convențională. Un studiu recent sugerează că, odată ce identificarea de către EM MALDI-TOF, același sediment poate fi utilizat pentru a efectua un disc de disc antibiograme, cu rezultate excelente, ceea ce permite reducerea în 24 de ore a studiului complet35. Schimbarea organizațională importantă că introducerea acestei metodologii presupune, iar infecțiile în care sunt diagnosticate câteva ore nu sunt critice pentru gestionarea pacientului, au făcut diagnosticul de infecție urinară prin intermediul emaldi-TOF nu a avut penetrarea pe care a avut-o, de exemplu, în cazul culturilor de sânge. Este necesar să se realizeze atât o sensibilitate mai mare, cât și o normalizare mai mare a metodelor de a lua în considerare introducerea lor în rutina clinică.
Identificarea directă a hemoculturilor
Stabilirea tratamentului empiric corect este decisiv în evoluția bacteriiemiei Probabilitatea ca tratamentul stabilit să fie corect, va fi mai mare cu cât informația este mai exactă și mai concretă pe care o putem furniza rapid. În acest aspect, posibilitatea oferită de EM Maldi-Tof de a obține o identificare fiabilă într-o perioadă scurtă de timp după pozitivizarea culturii sanguine este un avans evident.
Numeroase protocoale de prelucrare a sângelui-cultural au A fost descris (centrifugare diferențială, liza celulară și extragerea proteinelor prin metode chimice, separarea gelului …). Cele mai multe dintre ele se bazează pe liza și eliminarea componentelor celulare, fie prin centrifugare, cât și de spălări, în care compușii diferiți sunt utilizați ca saponină, clorură de amoniu sau SDS sau prin utilizarea tuburilor cu gel de separare seric și activator de coagulare. Pe scurt, ceea ce se caută cu aceste metode este de a izola și de a concentra microorganismele până la atingerea a cel puțin 105-107fc / ml, concentrația la care cantitatea de proteine este suficientă pentru a genera profiluri adecvate în emaldi-tof.
În cazul hemocolturilor există o procedură comercială aprobată (Seppsityper, Bruker Daltoniks GmbH, Germania), constând în adăugarea unei soluții de liză la o alicotă de 1 ml a culturii sângelui, după care eșantionul este supus la mai multe etape de spălare și centrifugarea și, în final, la o extracție convențională cu etanol și acid formic36.
Rezultatele procesării directe a culturilor de sânge sunt, în general, bune, cu procente de identificare corecte de 80-90%, deși cu unele nuanțe. Identificarea gram-negative este, de obicei, corectă în 90-95% din cazuri, în timp ce în gramoși este mult mai eterogenă, oscilarea între figurile similare cu cele ale gram-negative și cifre în jur de 50%. În cadrul acestora, ele prezintă probleme de fiabilitate asupra tuturor streptococilor grupului Viridans și a stafilococi non-coagulaze non-producătoare.
Rezultatele în cazurile de frangime, în primele studii, au fost dezamăgitoare. O extracție corectă, indispensabilă în acest caz, duce la figuri de identificare peste 90%, omologate cu cele obținute în bacteremias37. Este posibil să se ia în considerare posibil impactul anumitor componente ale unor hemoculturi, cum ar fi carbonul activ, care poate interfera cu identificarea. De asemenea, un timp de incubare prelungit al culturii sanguine poate avea un impact negativ asupra spectrelor obținute. Cu toate acestea, deoarece procedura obișnuită este de a efectua MS în cel mult câteva ore după pozitivizare, iar peste 80% din culturile semnificative de sânge pozitive sunt în primele 48 de ore, impactul clinic al acestui aspect nu pare să fie transcendent .
La evaluarea eterogenității procentelor corecte de identificare din hemoculturi, acesta trebuie luat în considerare faptul că nu există un criteriu omogen privind modul de evaluare a scorurilor obținute. Unii autori iau în considerare identificarea corectă cu valorile > 1.7. Alții diminuează această cerință de până la 1,5, dar includ cerințele precum identificarea se repetă în cele două sau trei poziții ale listei posibile de identificare sau că între primele două opțiuni oferite de diferența MALDI-TOF există o diferență de scor de scoruri cel puțin 0,3 puncte.
În general, nu suntem în favoarea reducerii nivelului de cerință în identificarea corectă în culturile de sânge, deoarece existența unui procent mai mare de „fără identificări” va presupune întotdeauna un risc mai mic, din punct de vedere clinic, decât proliferarea identificărilor incorecte, care ar putea condiționa tratamente inadecvate.
privind utilizarea metodelor de procesare comercială, cum ar fi Seppsityper, rezultatele în general sunt similare în ceea ce privește metodele de procesare manuală. Fiecare laborator trebuie să prioritiza între normalizarea și salvarea mai mare a timpului de procesare care a proprit aceste metode sau economii economice (aproximativ 1 EUR / eșantion) care implică utilizarea unei metode manuale.
în set, em maldi- TOF este o metodă rapidă și fiabilă pentru identificarea directă a microorganismelor în hemoculturi. Combinația cu metode care permit detectarea mecanismelor de rezistență la anumite antimicrobiene, permite să ofere informații clinice valoroase într-un timp semnificativ mai scăzut (24-48 ore mai mică), conform cerințelor metodologiei convenționale. Probabil principalul punct de elucidat, în acest moment, este modul de integrare a acestei noi activități în laboratoarele de microbiologie, astfel încât informațiile oferite sunt optimizate fără a presupune o supraîncărcare asupra serviciilor, de obicei nu cu precizie asupra tehnicianului de personal și cu structuri de timp foarte diverse.
Identificarea directă din alte eșantioane
Numărul de studii referitoare la aplicarea directă a emisiilor Mali-TOF către alte eșantioane biologice este mai mică. După cum sa discutat într-o altă secțiune, utilitatea EM MALDI-TOF în aceste eșantioane este determinată de mai mulți factori: pe de o parte, eșantionul trebuie să provină dintr-o zonă sterilă în condiții fiziologice și în care infecția, când apare, stochează pentru a fi de obicei monomicrobiene. Pe de altă parte, atât încărcătura bacteriană prezentă în eșantion, sunt esențiale, cum ar fi volumul de eșantion disponibil pentru studiu. Acest lucru face ca multe probe diferite de urină și hemocultură să prezinte probleme, de volumul scăzut disponibil (CSF, exudatele purulente), datorită concentrației scăzute de microorganisme (CSF, fluid peritoneal la pacienții supuși dializei peritoneale) sau prin prezența unor cantități importante de non – proteine bacteriene care pot modifica profilurile sau interferează cu ionizarea proteinelor bacteriene (exudatele purulente).
Sensibilitate scăzută în acele produse în care încărcătura bacteriană nu este foarte mare și volumul de eșantion disponibil, Deseori limitate, fac tehnici moleculare preferabile pentru diagnosticare rapidă, chiar și cu limitarea că gama de microorganisme detectabile într-un singur test este mult mai mică decât cu em cuzii -tof. Diagnosticele etiologice de meningită bacteriană au comunicat ocazional de CDRI-TOF pe CSF, dar un studiu raportat la 21erecmid a indicat că, pe 183 de eșantioane de CSF, dintre care 14 au fost pozitive pentru metoda convențională, nici unul nu a fost pozitiv de către em Cuzid-tof Direct Eșantionul38, care reafirmă faptul că, din punctul de vedere al sensibilității, Maldi-Tof Em nu poate concura cu tehnicile moleculare. În alte tipuri de exemple, experiența este foarte limitată, nu există protocoale sau date publicate privind sensibilitatea și specificitatea și, prin urmare, utilizarea clinică a emaldi-TOF, în acest moment, nu este pertinentă.
Media cromogenă
Deoarece apariția primului mediu cromogen (MC), cu mai mult de 30 de ani în urmă, a devenit un instrument foarte util pentru izolarea diferențială a microorganismelor patogene. În prezent există o mulțime MC comercializată pentru identificarea bacteriilor și a ciupercilor, precum și pentru studiul unor mecanisme de rezistență antimicrobiană.
fundamentul acestor mijloace în includerea unui substrat cromogen care, fiind hidrolizat de O enzimă specifică prezentă în microorganism, ea dă naștere unei colonii de colonare caracteristică, permite diferențierea sa.Ele sunt, de asemenea, mijloace selective, care inhibă creșterea altor microorganisme într-o cantitate mai mare sau mai mare, favorizând detectarea coloniilor colorate. Principalele substraturi ale MCS comerciale sunt derivatele indilice39 care pot fi hidrolizate de galactozidas sau glucozidaze, producând derivați toxici scăzuți și care nu inhibă creșterea bacteriană.
Detectarea microorganismelor gram-pozitive
Au fost dedicate mari eforturi mari. Dezvoltarea metodelor de detectare a meticilinei rezistente S. aureus (MRSA), având în vedere importanța acesteia în infecția nosocomială. În acest context, utilizarea MC a colectat o mare importanță pentru izolarea rapidă și identificarea acestei bacterii. Există numeroase MC pe piața detectării MRSA. Un studiu recent40 compară trei: CHROMID MRSA SMART (SMART), Generația CHROMID MRSA FIST (CHROMID) și strălucirea MRSA (OX2) pentru screening de 1.220 de eșantioane de pacienți spitalizați. Detectarea în aceste mass-media a fost coroborată de emaldi-TOF, diseminarea pe agar cu discuri cefoxitinei și PCR comercial pentru Mecca și MECC. Sensibilitatea la 24 ore a fost mai bună pentru mediul inteligent în raport cu mediul cromid, dar nu au existat diferențe semnificative cu mediul de ox2. Cu toate acestea, autorii indică faptul că este încă necesar, de a obține rezultate optime, îmbogățirea bulionului timp de 24 de ore înainte de inocularea în oricare dintre mass-media studiate.
Xu et al.41 Recenzii de la 5 din cele mai utilizate materiale media pentru cele mai utilizate. Detectarea MRSA. Cel care are ca rezultat cel mai bine acest studiu este strălucirea MRSA 2 (Oxoid Ltd, Termofisher, SUA), cu o sensibilitate de 65,7% și o specificitate de 99,8%. Randamentul acestui mediu se îmbunătățește dacă eșantionul este inoculat într-un bulion de îmbogățire înainte de ao planta în MC, cu o sensibilitate de 100% și o specificitate de 99,1%. Media cromagar MRSA prezintă o sensibilitate de 95% după 24 de ore de incubare, care ajunge la 100% dacă este incubată 48 de ore. Specificitatea în ambele cazuri este de 100%. Mediul BBL crom agar MRSA include cefoxitina în compoziția sa, ceea ce simplifică interpretarea rezultatelor. Acest mediu a fost studiat folosind izolatele MRSA obținute din hemoculturi, cu o sensibilitate și specificitate de 97,6 și, respectiv, 99%. MRSA SELECT a fost evaluat utilizând 652 izolate de hemoculturile după incubarea de 18-24 ore la 35 ° C, cu o sensibilitate de 99% și o specificitate de 98%, care a crescut cu 99% atunci când este combinată cu testul de coagulare. Cromediul mijlociu MRSA, care încorporează și cefoxitina în compoziția sa, a fost studiat în izolate din răniri și culturi de sânge. În hemoculturi arată o sensibilitate de 97,8% la 24 ore, ceea ce crește 100% la 48 ore și o specificitate de 99,7% care este menținută (99,6%) la 48 de ore. În răni, sensibilitate și specificitate au fost de 88,9 și 100% la 24 ore și 100% în ambele cazuri la 48 de ore.
Stabilirea comparațiilor între acest tip de mediu de cultură este complexă, deoarece există mulți factori, În afară de mediul de cultură, care poate influența rezultatele (tipul de eșantion, concentrația de inocul, timpul de incubare …). Cu toate acestea, datele disponibile și datele de specificitate sugerează că oricare dintre mediile menționate mai sus poate fi folosit ca o rutină pentru detectarea MRSA cu o fiabilitate ridicată.
Un alt grup de bacterii care au dobândit o importanță în infecție Nosocomial sunt. Enterococci rezistente la vancomicină (VRE), până la punctul în care CDC recomandă studiul transportatorilor în centrele de prevalență ridicate. Studiul transportatorilor VRE a fost realizat folosind Agar Sculin Bilis cu azidă și vancomicină (BEAV). Utilizarea sa a implicat o întârziere de diagnostic de 48 de ore, pe lângă necesitatea de a efectua teste complementare pentru identificarea corectă. Pentru a accelera acest proces, au fost dezvoltate diferite MCS. Un studiu recent compară cinci mc pentru VRE utilizând 400 de probe scaun. Mijloacele utilizate au fost Intray Colorx VRE (BioMeddiagnostics, oraș alb, sau), cromid vre (BioMérieux, Marcyl’étoile, Franța), VRESELECT (Bio-Rad, Marnes-La-Coquette, Franța), Hardychrom Vre (Diagnosticare Hardy, Santa Maria, CA) și Spectra VRE (Remel, Lenexa, KS), folosind Agar Agar și bulionul Beav ca metodă de referință. Citirea a fost efectuată la 24 de ore. Rezultatele celor cinci mass-media arată o sensibilitate între 89,9 și 94,9%, mai mare în toate cazurile decât cele indicate de Agar Beav (84,9%). Cele mai bune date de sensibilitate și specificitate continuă să le ofere BEAV, dar în detrimentul unei întârzieri de diagnosticare de până la 48 ore în raport cu MC. Cel mai sensibil MC din acest studiu a fost cromid VR (94,9%), deși fără diferențe semnificative statistic cu restul.
Specificul și capacitatea de a diferenția speciile de enterococcus au fost similare pentru cele cinci MC. Intray Colorx VR a avut cea mai mică specificitate cu 98,8%, în timp ce celelalte patru plăci au fost de 99,7%. Caracteristicile de sensibilitate și specificitate ale acestor mijloace o fac recomandată pentru detectarea transportatorilor vre atunci când acest lucru este pertinent. Ele sunt, cel puțin, perfect comparabile cu agarul Beav, scăderea timpului de detecție la 24-48 ore.
Detectarea microorganismelor gramatice
în 1979, kiliană și lovitură descriu un nou mediu de cultură care utilizează β-glucoronidaza ca a Substrat pentru detectarea directă a E. coli în uroculturi. Studiile de rentabilitate au indicat că această metodă a implicat o economie economică de 46% și o reducere de timp de identificare de 64% în ceea ce privește metodele convenționale39. De atunci, numeroasele MCS au fost dezvoltate cu substraturi diferite pentru identificarea principalilor uropatogeni. Unul dintre mass-media comercializat recent este Elite CPS Chromid (BioMérieux, Durham, NC) care permite identificarea directă a E. coli și identificarea prezumtivă a enterococcus spp., A unor enterobacteriaceae și a bacteriilor grupului PROSAE, deși în aceste cazuri Confirmarea identificării este necesară prin teste biochimice sau emaldi-tof.
Eficacitatea acestui mediu pentru detectarea urroopatogenentului43 a fost evaluată, evaluând timpul necesar pentru diagnosticarea și consumabilele utilizate în comparație cu Utilizarea agarului de sânge și a agarului MacConkey. Concordanța acestui mediu cu metodologia convențională a fost de 88% pentru urină semnificativă din punct de vedere clinic, de 74% pentru urină, cu o creștere nesemnificativă, 69% pentru contaminat și 95% pentru plăci fără creștere bacteriană. Principalele discrepanțe au fost cauzate de creșterea agarului de sânge, dar nu în MC de bacterii Grampositivas, în general, cu puțină relevanță clinică în uroculturi, cum ar fi Staphylococcus spp. și lactobacillus. În ceea ce privește timpul scurs între însămânțarea eșantionului și identificarea coloniilor, diferența nu a fost semnificativă statistic dacă cele două mijloace sunt comparate la nivel global (27,2 ore din media pentru metoda convențională comparativ cu 26,6 ore pentru MC), dar a fost în Acele urinari în care E. coli au crescut în cultura pură (27,1 ore în metoda convențională comparativ cu 24,4 ore în cromid, p
0,0001). Autorii susțin că acest timp de îmbunătățire se datorează ușurinței și fiabilității identificării culorilor, care evită necesitatea unor metode complementare de identificare.
CPS Media Chromid ™ (CPS4) (Biomérieux, St. Laurent, QC) și UrisELECT ™ 4 (URS4) (Bio-Rad, Montreal, QC) au fost evaluate44, peste 903 de probe de urină, comparativ și cu metoda convențională de agar de sânge și cu agar MacConkey. Concordanța cu metoda convențională a fost de 89,3 și 89,5% pentru URS4 și respectiv CPS4. Dacă numai probele în care creșterea a fost considerată semnificativă din punct de vedere clinic, această concordanță a crescut la 93% în cazul URS4 și 93,1% pentru mediul CPS4.
În toate cazurile, autorii sunt de acord că cel mai mare avantaj al lui Utilizarea MC este raționalizarea procesului de identificare, prin reducerea nevoii de teste complementare, care permite mai repede un diagnostic etiologic.
Având în vedere utilitatea demonstrată din acest instrument de diagnosticare, mai multe grupuri proiectează noul MC , unii nu au fost încă comercializate, ceea ce ar putea presupune o alternativă interesantă în viitor. Un nou media45 a fost proiectat pentru detectarea bacteroidelor, care include în formularea sa 3,4-ciclohexanoscuzu-p-D-glicozide, care este ținta acțiunii de bacteroizi și care dă naștere la aspectul colonii de negru. Acest mediu a fost comparat cu agarul bilis sculinar în 100 de probe fecale. MC a permis identificarea lui B. Fragilis în 34 de eșantioane, în timp ce în Agar Bilis Esculina, acest microorganism a fost detectat în 19 mostre. În plus, MC a fost mult mai selectiv, deoarece în nici un caz nu a fost o creștere a speciilor non-sexe, în timp ce în Agar Bilis Esculina a fost observată creșterea microorganismelor aparținând altor genuri (Enterobacteria, Clostrium, Candida …) cu 34 de ocazii. Autorii studiază, de asemenea, capacitatea acestor MC, suplimentată cu meropenem și cu metronidazol, pentru a detecta prezența bacteroidelor rezistente la unul sau alt antimicrobian, demonstrând că în ambele cazuri sunt capabile să detecteze tulpini rezistente cu o sensibilitate de 100% și a specificitate de cel puțin 85%.
Un alt mediu inovator dezvoltat este cromagarul enterocolicy yersinia care prezintă ca un avantaj față de mediul de cesssulodină-irgasan-novobiocin, posibilitatea creșterii tensiunii Y. enterocolitica Serovar O3 și Y. Psedotuberculosis, a cărui creștere poate fi văzută inhibat în mediu CIN46.
Detectarea drojdiei
Deși primul MC dezvoltat pentru ciuperci, la începutul anilor 90, au avut puțin succes în aplicarea lor clinică prin capacitatea lor limitată de diferențiere, MC pentru micologia dezvoltată în ultimii 15 ani au avut un succes mult mai mare, deoarece permit o identificare rapidă prezumtivă a diferitelor specii de drojdie, un studiu mai bun al culturilor mixte și identificarea timpurie a speciilor asociate cu rezistența antifungică. Mijloacele dezvoltate se bazează, ca în cazurile anterioare, în prezența substraturilor pentru una sau mai multe enzime (β-N-acetil hexosaminidază și, în unele cazuri, β-glucozidază sau fosfatază), care permit o identificare rapidă, cel puțin, De Candida Albicans, și în numeroase cazuri identificarea definitivă sau presupusă a Candida Gabrada, Candida Lusitaniae, Candida Kefyr, C. Parapsiloza, Candida Tropicalis și Candida Krusei. Studiile disponibile și experiența deja prelungită cu acest tip de mass-media demonstrează o specificitate ridicată de specificitate și o bună sensibilitate. Cu toate acestea, având în vedere variabilitatea culorii și a morfologiei care pot apărea în izolatele aceleași specii, identificarea ar trebui să fie întotdeauna considerată presupusă. Un studiu recent privind mai mult de 5.600 de eșantioane clinice pozitive pentru drojdie, arată că mai mult de 8% au fost culturi mixte, a căror detecție este mult mai probabilă dacă MC este utilizat și propune un algoritm care combină MC și EM Maldi-TOF ca procedură mai adecvată de identificare47.
Conflictul de interese
Autorii declară să nu aibă nici un conflict de interese.