Masses Spectrometry Maldi-TOF. Origins
A introdução da espectrometria de massa (MS) A dessorção a laser assistida por matriz / ionização do voo (Maldia-TOF) tem sido, com toda a probabilidade, a mudança tecnológica do maior projecto aconteceu na clínica de microbiologia no última década. Em poucos anos, passou de ser uma novidade promissora, para ser uma tecnologia totalmente integrada na atividade clínica diária e disponível, em nosso país, nos serviços de microbiologia de numerosos centros hospitalares1. Para isso, foi necessário que os espectrômetros de massa sofrem uma evolução tecnológica significativa. A este respeito, havia dois progressos que eram cruciais. Primeiro, o desenho do sistema de voo (TOF) por w.e. Stephens, em 1946, que permitiu a separação de diferentes massas. Ao acelerar os íons em um campo elétrico e adquirir toda a mesma energia cinética, a velocidade que adquire e, portanto, o tempo usado para viajar o tubo de vácuo, depende da massa da molécula ionizada, que pode ser inferida a partir do tempo gasto Esta rota.
O segundo marco foi o desenvolvimento de métodos que permitiram proteínas intactas ionizantes que, devido ao seu tamanho, até então não tinham sido suscetíveis à ionização, uma vez que a alta energia necessária para ele acabou alterando ou destruindo sua própria proteína. Em 1987, Köichi Tanaka apresenta um novo método de análise (dessorção a laser suave) que permitiu a transferência para as moléculas a energia necessária para ionizá-las sem quebrar os frágeis títulos químicos. Esta descoberta lhe valeu o Prêmio Nobel de Química em 2002, juntamente com John B. Fenn, para “o desenvolvimento de métodos de identificação e análise estrutural de macromoléculas biológicas”. Não havia mais espectrômetros de massa baseados nesse tipo de análise, embora algumas mudanças fossem necessárias no método para que o EM MALDI-TOF acabasse, em que a dessorção dos íons é favorecida por uma matriz que absorve a energia do laser e parcialmente transferi-lo para o objeto de moléculas de estudo. Mesmo que essas modificações melhorassem a técnica, tornando mais fácil e fácil, os responsáveis por essas melhorias, Michael Karas e Franz Hillenkamp, não foram incluídos no Prêmio Nobel, que gerou uma importante controvérsia. Com base nas contribuições desses autores começaram a ser possíveis, o estudo de macromoléculas e biopolímeros por MS, que abriu novos campos de aplicação para esta tecnologia.
em seus quase 30 anos de vida, os Maldi em – TOF foi usado para a análise quantitativa e qualitativa de proteínas de várias origens. Inicialmente, foi aplicado a proteínas anteriormente isoladas ou pequenos conjuntos de proteína, mas graças aos avanços técnicos tanto na instrumentação quanto nas ferramentas para a análise de computadores dos dados, o estudo de grandes grupos de proteínas agora pode ser abordado.
A possibilidade de começar a estudar proteínas complexas, e não apenas peptídeos pequenos, amplamente expandiram os potenciais campos da aplicação de MS. Um deles foi a identificação de microorganismos que, como o procedimento foi simplificado e o software necessário foi melhorado para explorar os dados brutos fornecidos pelo espectrômetro, rapidamente derivado em sua aplicação clínica.
Mass SpTtrometric Malzi-TOF em microbiologia clínica
Até a introdução de EM MALDI-TOF, a identificação bacteriana, mesmo com avanços significativos, como a criação de galerias de identificação miniaturizada e a automação de sua inoculação e leitura, continuaram a ser alimentadas pelos métodos desenvolvidos por bacteriologia A prática dos sistemas de identificação continuou a basear-se na fermentação de açúcares e sua detecção através da mudança de pH gerada, a metabolização de outros substratos e a produção de diferentes metabólitos e atividades enzimáticas detectáveis por métodos químicos. Todos estes métodos adoliram várias limitações:
- •
- •
mostrou problemas de identificação em todos os microrganismos com dificuldades para crescer na mídia líquida utilizada para a inoculação desses painéis, bem como em microrganismos com baixa atividade bioquímica e enzimática.
- •
foi necessário considerar a margem de erro derivada do fato de que, indivíduos da mesma espécie podem ter diferentes comportamentos contra vários substratos.
O crescimento bacteriano requerido, que supõe, na maioria dos casos, um período de incubação de pelo menos 16-18 h de sua inoculação até a leitura.
Essas limitações, embora fossem conhecidas e assumidas, elas se tornaram mais claramente patentes quando, dadas as discrepâncias observadas em alguns estudos entre o MSZ-TOF e a identificação convencional, O sequenciamento dos 16 anos mostrou que, na grande maioria dos casos, a identificação correta correspondia à fornecida pelo EM Maldia-Tof2.
A este respeito, o EM MALDI-TOF tem vantagens óbvias:
Independentemente da possibilidade de identificar microorganismos diretamente de algumas amostras, que é tratado em outra seção, até mesmo o crescimento de placa muito escassa ou precoce permite uma identificação confiável em um curto período de tempo , pelo menos, pelo menos, aqueles 16-18 h de crescimento nos sistemas de identificação bioquímica.
A análise do perfil de proteína do microrganismo no espectro do 2- 20kd, que é onde a maioria das proteínas de costela são colocadas OSÔNICO, oferece para a grande maioria das espécies bacterianas Um perfil específico, que permite a diferenciação do resto com uma confiabilidade semelhante à oferecida pelo seqüenciamento dos 16s RNR.
A isso deve ser adicionado que, a introdução desta tecnologia expandiu grandemente a gama de gêneros e espécies que somos capazes de identificar com confiabilidade, com um método capaz de ser usado em rotina. Isso até levou a uma reavaliação do papel como patógenos de microorganismos que, devido à dificuldade de sua identificação pelos métodos clássicos, eram mais prováveis infradiagnosed3.4.
em 1975, Anhalt e Fenselau propõe o uso de EM para a identificação de microorganismos5. Vinte anos depois, o primeiro estudo é publicado que demonstra a eficácia do Maldia-TOF EMS para a identificação de microorganismos a partir de células completas6.
em 2009 foi publicado que provavelmente foi um artigo fundamental para saber , especialistas envolvidos no diagnóstico de doenças infecciosas, as possibilidades de em Maldia-TOF7. Estuda mais de 1600 isolados que incluem bactérias grampoositivas e gramnegativas, aeróbicas e anaeróbias, obtendo 95,4% das identificações corretas. Este artigo destina-se a este artigo que foi então transcendido para a utilidade prática desta metodologia: a disponibilidade de bases de dados de microrganismos suficientemente amplos, tanto do ponto de vista qualitativa (número de gêneros e espécies incluídos) e quantitativos (os autores demonstram Que a probabilidade de identificação correta é maior para os microorganismos que existem pelo menos dez perfis diferentes introduzidos no banco de dados).
A partir deste momento, a expansão rápida de uso ocorre dessa tecnologia na microbiologia clínica. A primeira publicação na Espanha ocorre em 2010, mostrando uma correlação com a metodologia convencional, a nível de espécies, 100% em gramíneas e 87,7% no gramnegativo8. Desde então, o número de publicações relacionadas a diferentes aspectos do uso clínico de EM Maldia-TOF, mas acima de tudo à identificação de microorganismos, foi exponencial7-12.
Identificação de bactérias gramnegativas usando Maldia-TOF
Estudos mostram que a eficiência na identificação de Enterobactérias e outros gramas-negativos é excelente, incluindo os mais comuns gramas-negativos em clinicamente e outros menos frequentes ou mais complicados para identificar com metodologia clássica (espécies diferentes de Yersinia, Aeromonas, Plasiomonas ,. Brucella, Francisella, Achromobacter, Stenotropomonas, Burkholderia …). Como em outros casos, os problemas de identificação quase sempre foram ligados a insuficiências de bases de dados, em vez de limitações do método, como ocorreu em alguns estudos com gêneros como Ralstonia, Elizabethkingia ou Sphyngobacterium13. Inicialmente, algumas limitações foram levantadas que poderiam ter maior transcendência clínica, como a incapacidade de diferenciar entre Escherichia Coli e Shigella14 ou a dificuldade de diferenciar serovares de Salmonella Enterica. Estudos mais recentes sugerem que é possível salvar essas limitações. Usando programas de computador, como flexanalisia e clinprools (Bruker Daltonics GmbH, Alemanha), picos específicos foram identificados que permitem diferentes de E. coli e shigella em 90% dos casos15. Outros autores mostraram que essa diferenciação é possível mesmo sem usar qualquer software adicional, simplesmente aumentando o número e a variedade de perfis de E. coli e Shigella presentes no banco de dados de referência16. Com este método, os autores identificam corretamente os isolados 60/64 dos isolados de E. coli e 110/116 de Shigella.
Para todos aqueles que usaram o EM MALDI-TOF para identificação, consistiu que, com a metodologia usual, a identificação de salmonela em um nível de gênero é confiável, mas além do nível de gênero é muito menos. No entanto, já existem publicações de 2004 que sugerem a existência de picos específicos que poderiam permitir identificar serovares com maior confiabilidade17. Um estudo recente sugere a existência de picos que permitem identificar de forma confiável a Typhi18 serovar. Como os elementos diferenciais parecem existir, uma extensão do banco de dados de referência com um maior número de alto-falantes, pelo menos, os serovares mais frequentes, provavelmente melhorariam a resolução neste caso particular.
em outros casos, Como é a diferenciação entre espécies do complexo Cloacaee Enterobacter, a capacidade do EM MALDI-TOF é de cerca de 80%, que sem ser ideal, representa uma melhoria sensível em relação à metodologia convencional19.
no Outras mãos, como já mencionado, a identificação correta de microorganismos previamente identificadas incorretamente, como o gênero Raoultella, freqüentemente identificada por métodos clássicos, como Klebsiella ou como Enterobacter, está permitindo uma visão mais real do seu papel como patógenos, anteriormente subvalorizados3.
Identificação de Bactérias Grampaitositivas usando Maldia-Tof
A identificação de microorganismos Gram-positivos geralmente mostram figuras mais baixas, devido Em alguns casos para a dificuldade de mentir a parede, e em outros à semelhança entre espécies. Em geral, o método direto (aplicação da colônia junto com a matriz na placa do espectrômetro) é geralmente suficiente para obter uma boa identificação da colônia, mas às vezes a extração anterior com etanol / ácido fórmico pode ser necessária para obter valores de identificação ideais. Juntos, oferece excelentes resultados na identificação de Staphylococcus aureus e outras espécies e subespécies de staphylococci, tanto produtores como não produtores de coagulase, bem como de enterocci e outras mortes menos frequentes como microcócitos, Gemella, Rothia, etc. Também demonstrou sua utilidade na identificação de bacilos gram-positivos, incluindo alguma identificação complexa por métodos clássicos (Listeria, Lactobacillus, Corynebacterium, Nocardia, Actinotignum …) 20.
Além disso, como em O caso de gramados, o uso sistemático desta tecnologia está mudando de consideração como patógenos de alguns microorganismos, como Staphylococcus lugdunensis21. Juntos, os dados disponíveis tornam aconselhável, no caso de Staphylococci para uso, como em qualquer outro caso, os bancos de dados que oferecem um maior número de espécies e um número razoavelmente alto de perfis por espécies, e usam um método de extração no lugar de Aplicação direta, bem como aplicar o microrganismo na placa em duplicado. A redução da pontuação necessária para validar uma identificação de nível de espécie para valores > 1.7, em vez de > 2.0 como é comum , como alguns autores22 propõem, podem aumentar o número de identificações no nível da espécie, mas em troca de uma diminuição na especificidade cuja relevância pode ser discutível.
Provavelmente, o grupo de estreptococos é o que apresenta maiores problemas entre gramsitivos. Enquanto algumas das principais espécies patogênicas (Streptococcus pyogenes, estreptococcus agalactiae) são identificados com alta confiabilidade, Streptococcus pneumoniae (S. pneumoniae) sempre foi uma fonte de problemas, devido à sua confusão com outras espécies do grupo de Mitis. As principais equipes em uso em nosso país identificam S. pneumoniae com confiabilidade. O erro mais comum é a identificação errada de outros estreptococos do grupo de Mitis, como S. pneumoniae, especialmente com o banco de dados do Bruker23. Alguns picos foram descritos como S. pneumoniae, então alguns autores recomendam seu uso para corroborar essas identificações23. No entanto, a última biblioteca de referência da Bruker Daltonics (MBT 6903 MSP Library) continua a recomendar o uso de outros testes (optoquina, solubilidade na bile) para diferenciá-los.
Mesmo dentro do mesmo grupo, a qualidade de A identificação das diferentes espécies pode variar consideravelmente, como ocorre dentro do grupo AnginoSus StrepTococcus.
Em qualquer caso, como no Staphylococci, o uso de um banco de dados tão amplo e completo possível, e a identificação em duplicada e com protocolos que incluem extração são recomendados para otimizar os resultados (Fig. 1).
Grampositive, grama-negativo, anaerobi e processamento de levedura usando Maldia-Tof.
Identificação de bactérias Anaerobias através de Maldia-TOF
A identificação de bactérias anaeróbicas, grebous e gramnegativa, tem um alto grau de confiabilidade, identificando corretamente a espécie mais comum (bacteróides, precipotella, porfiromonas, fusobacterium, clostridium, actinomyces …). A aplicação do EM MALDI-TOF neste domínio também mostrou que a identificação bioquímica foi muito menos confiável do que pensada3. A combinação de simplicidade, rapidez e confiabilidade torna a EM MALDI-TOF no método de escolha para a identificação de rotina de bactérias anaeróbias na clínica. A extração ou não a amostra não parecem modificar significativamente os resultados, nem faz o meio de cultura de origem. Um fator decisivo, como em outros casos, é a amplitude dos bancos de dados. Já um dos primeiros estudos realizados na Espanha3 mostrou que a maioria dos erros na identificação de bactérias anaeróbicas deveu-se à ausência de perfis de referência da espécie correspondente no banco de dados. Estudos mais velhos oferecem uma eficácia na identificação em torno de 75-80%, enquanto estudos mais recentes, com bancos de dados mais completos ou bancos de dados específicos oferecem números acima de 90%. A capacidade de identificar ribotipos difficile Clostridium, sugeridos por alguns autores, não foi definitivamente demonstrado.
identificação de fungos de levedura e filamentosos através de Maldi-Tof
A identificação de levedura foi mostrada a partir dos excelentes resultados, diferenciando mesmo espécies complicadas de discriminação por métodos convencionais, como o complexo de parapsilose candida. No entanto, os resultados em relação aos fungos filamentosos foram muito mais erráticos. Alguns estudos iniciais mostraram bons resultados seleção seletivamente esporos, mas esse método é impraticável em um laboratório clínico, então a maioria dos estudos foi direcionada ao estudo conjunto de esporos e hifas. No caso de fungos, a extração convencional com ácido fórmico e acetonitrilo melhora significativamente os resultados. Outros métodos que foram testados, como o uso de contas de vidro para fragmentar as paredes, não parecem oferecer resultados significativamente superiores à extração convencional.
Um problema colocado por fungos filamentosos é a existência de diferenças significativas Nos perfis de proteína obtidos de acordo com a antiguidade das culturas, e mesmo entre a subcultura diferente do mesmo CEPA24. A solução para isso passa pela elaboração de bancos de dados mais amplos e complexos, que incorporam perfis de um maior número de cepas e culturas de diferentes antiguidades. Alguns fabricantes recomendam a remoção após uma safra de um dia em caldo, embora isso tenha um impacto em um atraso de 24 h na emissão do resultado. Um estudo realizado em 20125 mostra que uma base de dados bem elaborada e suficientemente complexa é fundamental, e que pode melhorar a identificação de fungos filamentosos a figuras semelhantes às obtidas com bactérias e leveduras. Infelizmente, a elaboração e a validação desses bancos de dados são complexas e não estão disponíveis para muitos usuários, e poucos desenvolvidos estão disponíveis. Portanto, e também para fins de padronização, é provavelmente mais apropriado usar os bancos de dados dos fabricantes, que devem ser convenientemente expandidos.
Atualmente Bruker Daltonics, cuja base de dados de fungos filamentosos, na sua versão MBT 6903 MSP Library , Inclui 25 gêneros e 42 espécies, recomenda a cultura em meio líquido durante a noite, seguida de centrifugação, lavagem e extração com etanol, ácido fórmico e acetonitrilo (Fig. 2). O MS-Vitek, em sua versão 3.0, inclui 32 gêneros e 81 espécies, e Saramis em sua versão Ruo 4.13, inclui um banco de dados mais amplo, com 45 gêneros e 168 espécies. A Biomériieux não recomenda cultura em meio líquido, mas uma extração convencional com etanol, ácido fórmico e acetonitrilo (Figura 2). Apesar de tudo, um estudo recente utilizando a metodologia recomendada por Bruker identifica corretamente, a nível de espécie, apenas 72% do insulte26, e um estudo muito recente com o Vitek 3.0 identifica corretamente 66,8% de 318 isolado, devido a do banco de dados27. Portanto, hoje, a identificação de fungos filamentosos por EM Maldi-TOF ainda não pode substituir completamente a metodologia de identificação convencional.
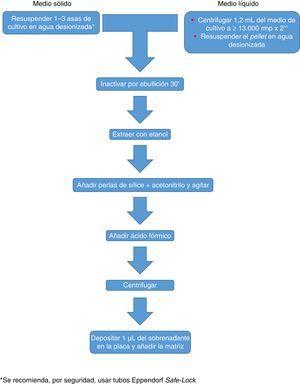
Recomendado processamento mycobacteriano para o sistema Biotyper Maldi.
div id = “4895790f6a”> Identificação de micobactérias usando maldi-tof
as limitações da metodologia tradicional para a identificação de micobactérias, em tudo em que se refere ao tempo de resposta, eles tornaram a totalidade prática dos laboratórios decantados pelos métodos de identificação molecular. O uso de EM MALDI-TOF para a identificação de micobactérias é uma alternativa igualmente rápida e mais barata do que as técnicas moleculares, mas mostra peculiaridades em vários aspectos. Por um lado, os métodos diretos de análise, ou que envolvem uma alta concentração ou manipulação dos microrganismos anteriormente à sua inativação, não são recomendados por uma questão de segurança biológica. O uso de um método de extração que garante a ruptura das células melhora a qualidade dos espectros e aumenta a segurança do procedimento.
Vários fatores podem influenciar a eficácia da identificação da Mycobactérias por Em Maldi – TOF. Como também foi mostrado com fungos, micobactérias pode ter perfis de proteína muito diferentes, dependendo da antiguidade das culturas, por isso é importante para o diagnóstico incluir perfis de proteína de culturas de diferentes antiguidades para cada microrganismo. Também foi falado sobre a possível formação de polímeros que mastigam o tamanho real das proteínas características usadas para identificação. Tudo isso torna a identificação de micobactérias um procedimento menos previsível em termos de seus resultados do que os de outras bactérias, como demonstra a heterogeneidade de dados publicados. Nas últimas atualizações, as bibliotecas experimentaram melhorias significativas. Assim, por exemplo, a biblioteca micabacteriana de Biotyper 3.0 inclui 149 espécies, e a versão 3.0 do Vitek MS incorpora 48 novas espécies em relação à versão anterior.
O procedimento inicialmente recomendado por Bruker incluiu uma série de etapas (ajustando a Uma certa densidade óptica, várias etapas de centrifugação e ressuspensão) que assumiram um risco biológico provavelmente desnecessário, então eles foram substituídos por um único passo de tratamento com calor. O método recomendado inclui atualmente vários passos de aquecimento e lavagem, seguidos por um tratamento com bolas de sílica e acetonitrilo. Posteriormente, eles são tratados com ácido fórmico, aplicado na placa e cobertos com a matriz (Fig. 3). Pelo contrário, o método defendido pela parte biomérieux de um tratamento inicial com 70% de etanol e bolas de sílica, após o qual a amostra é extraída com ácido fórmico e acetonitrilo, aplicado na placa e coberta com a matriz (Fig. 4). / p>
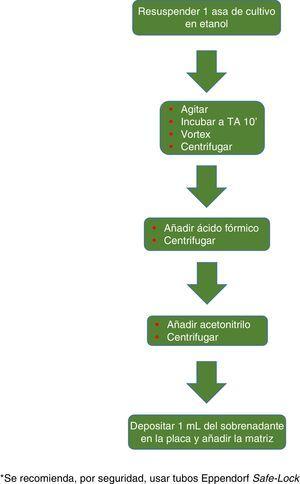
Recomendado processamento mycobacteriano para o sistema Vitek MS.
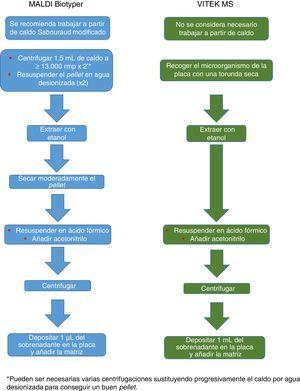
processamento fungal recomendado para sistemas MALDI Biotyper e Vitek MS.
um estudo comparativo entre os dois métodos realizados recentemente NTE28 mostra que, embora a identificação tenda a ser algo melhor a partir de metade 7h10 do que a Lowenstein Jensen, as diferenças são pequenas em ambos os sistemas. No entanto, demonstra que o método de processamento da amostra é crítico, e não é intercambiável entre os sistemas. As amostras obtidas com o método de extração brutador proporcionam melhores resultados em seu espectrômetro e o processado com o método Vitek fazê-lo com Salamis e Vitek MS, embora a diferença neste caso seja menor. Juntos, a Vitek MS parece ser menos afetada pelo método de extração. Em relação à eficiência na identificação, Bruker Biotyper identificou uma pontuação ≥1,8 81,5% dos isolados, enquanto Saramis e Vitek MS 3.0 identificou com uma confiabilidade ≥90% em 85,4 e 89,2%, respectivamente. Usando estes mesmos critérios, 22,6% dos isolados que foram identificados por Bruker Biotyper precisavam ser extraídos em mais de uma ocasião para alcançar a identificação, que ocorreu 21,5% com saramis e 14,2% com VITEK MS 3.0.
Em relação a outros microrganismos com estrutura parietal próxima à da micobactéria, por tempo, tem sido considerado que sua identificação era problemática devido à composição de sua parede. No entanto, se foi demonstrado que, se um banco de dados estiver disponível com referências suficientes, um método de extração semelhante ao usado com micobactérias pode oferecer bons resultados29.
Todos os dados referidos acima sugerem que o espectro de microorganismos suscetíveis A identificação por EM Maldia-TOF é extraordinariamente ampla e que, com exceções contabilizadas, o fator limitante da capacidade do Maldia-TOF emuses quase sempre mais na integralidade dos bancos de dados de referência do que na capacidade do método para obter perfis confiáveis de quase qualquer microrganismo bacteriano capaz de crescer em meios de cultura. De fato, as empresas de fabricação parecem estar cientes do problema; Assim, a última biblioteca de referência publicada por Bruker Daltonics (MBT 6903 MSP Library) incorpora 938 novos perfis, dos quais 17,2% visam cobrir novos gêneros e espécies (15 novos gêneros e 96 novas espécies especificamente), em particular anaeróbios e fungos, Mas 82,8% visam aumentar a diversidade de perfis dentro das espécies já contempladas em bibliotecas anteriores. Da mesma forma, a VITEK MS versão 3.0 inclui 242 novas espécies bacterianas, entre as quais são 48 novas Micobactérias e 15 noites, e 55 novos fungos, com uma média global de mais de 10 espectros isolados e 25 espectros por espécies.
Identificação de microorganismos, por Maldia-TOF, da amostra direta
A possibilidade de identificação de microorganismos antes do crescimento das colônias tem sido, desde o início, um objetivo muito atraente, então suposta o tempo de poder estabelecer um Tratamento empírico mais orientado, reduza o surgimento da resistência e otimize as despesas30. O EM MALDI TOF, aplicado à amostra direta, tornou-se uma ferramenta muito útil em infecções graves com alta taxa de morbidade, como bacteremia e fungemias, embora o trabalho direto em amostras tenha inconvenientes que possam limitar a utilidade da técnica. Em amostras com baixa densidade microbiana, não é possível realizar uma identificação correta, uma vez que a quantidade de proteínas bacterianas presentes é insuficiente. Algo semelhante ocorre com as amostras poliméticas, uma vez que um espectro de proteína aberrante pode ser gerado, como resultado da mistura de vários perfis, ou ignorando diretamente o microrganismo que está em uma proporção menor. Algumas melhorias de software foram desenvolvidas, que às vezes podem detectar e até mesmo identificar os microorganismos envolvidos separadamente, mas requer ser cuidadosamente estudado e provavelmente refinado a ter utilidade real. Na maioria dos métodos que funcionam diretamente na amostra, e antes da possível presença de microorganismos intracelulares, é aconselhável usar métodos que incluem lise celular.
nem todas as amostras são válidas para este tipo de estudo. A amostra ideal é aquela que vem de uma área habitualmente estéril, que pode acomodar altas concentrações de microrganismo e nas quais não há limitações significativas em relação ao volume de amostra. Amostras de áreas habitualmente colonizadas (pele, fezes, altos aparelhos respiratórios …) gerará perfis aberrantes em toda a probabilidade. Uma questão técnica para levar em conta ao trabalhar diretamente na amostra é o volume de amostra disponível, que em casos como urina ou culturas sanguíneas geralmente não é um problema, mas em outros com fluido cerebrospinal, e que será como muito mais importante que menor é a concentração bacteriana. Além disso, amostras com alto teor de proteína de origem alienígena para o microrganismo também podem gerar problemas no momento de sua interpretação31.
No entanto, a vantagem que é, especialmente nas hemoculturas, ultrapassa 24 h a identificação de O microrganismo e orientação do tratamento mais especificamente, fez com que o uso direto de MS na amostra, especialmente nesses casos, tenha generalizado.
Identificação direta das amostras de urina
Urina é uma das amostras que melhor se ajustam ao Condições ideais para trabalhar com EM Maldi-TOF na amostra direta. É um fluido estéril sob condições fisiológicas, a carga bacteriana na urina infectada é alta na maioria dos casos, e as limitações relativas ao volume de amostra e obtenção de novas amostras, se necessário, são mínimas.
Estudos com excelente Os resultados foram publicados, com concordância na identificação que atingem 90-95% 32,33 em comparação com sistemas automatizados.Diferentes protocolos de processamento de amostros foram descritos, todos eles coincidindo na necessidade de uma pré-triagem (citometria de fluxo, manchas de grama) para discriminar entre amostras positivas e negativas e limitar o uso de MS a exemplos inicialmente positivos34.
Com pequenas variações, a preparação das amostras geralmente consiste em submetê-las a uma série de centrifugações e lavada com água desionizada, então realizando o procedimento convencional da Maldia-TOF. As modificações foram feitas pela incorporação da alíquota da amostra SDS a 10% 33 para melhorar a liberação de proteínas, ou Tween-8034 para melhorar os resultados. Os resultados obtidos são geralmente melhores com gramado do que com grampopositives e fungos, melhorando a confiabilidade quando as contagens são altas, acima de 105fc / ml.
Um inconveniente deste método tem sido a necessidade de realizar o estudo de sensibilidade através da metodologia convencional. Um estudo recente sugere que, uma vez que a identificação por EM MALDI-TOF, o mesmo sedimento pode ser usado para realizar um disco de disco antibiograma com excelentes resultados, o que permite reduzir em 24 horas o estudo completo35. A importante mudança organizacional que a introdução desta metodologia supõe, e infecções em que, muitas vezes, algumas horas são diagnosticadas não são críticas para a gestão do paciente, fizeram o diagnóstico de infecção urinária através de em Maldi-TOF não teve o penetração que teve, por exemplo, no caso de culturas sanguíneas. É necessário atingir uma maior sensibilidade e uma maior normalização dos métodos para considerar sua introdução na rotina clínica.
Identificação direta de hemoculturas
O estabelecimento de tratamento empírico correto é decisivo na evolução da bacteriemia A probabilidade de que o tratamento estabelecido esteja correto, será maior quanto mais precisas e concretas as informações que pudermos fornecer rapidamente. Nesse aspecto, a possibilidade oferecida pelo EM MALDI-TOF de obter uma identificação confiável em um curto período de tempo após a positivização da cultura do sangue é um avanço evidente.
Numerosos protocolos de processamento sanguíneo foi descrito (centrifugação diferencial, lise celular e extração de proteínas por métodos químicos, separação de gel …). A maioria deles é baseada na lise e na eliminação de componentes celulares, seja por centrifugações e lavagens, no qual diferentes compostos são usados como saponina, cloreto de amônio ou SDS, ou usando tubos com gel de separador sérico e ativador de coagulação. Em suma, o que é buscado com esses métodos é isolar e concentrar microorganismos até atingir pelo menos 105-107FC / ml, concentração ao qual a quantidade de proteínas é suficiente para gerar perfis adequados em Maldia-TOF.
No caso de hemoculturas existe um procedimento comercial aprovado (SEPSityper, Bruker Daltoniks GmbH, Alemanha), consistindo da adição de uma solução de lise para uma alíquota de 1 ml da cultura sanguínea, após a qual a amostra é submetida a várias etapas de lavagem e centrifugação e, finalmente, em uma extração convencional com etanol e ácido fórmico36.
Os resultados do processamento direto de culturas sanguíneas são geralmente bons, com percentagens de identificação corretas de 80-90%, embora com algumas nuances. A identificação de grama-negativos é geralmente correta em 90-95% dos casos, enquanto em gramsitivos é muito mais heterogênea, oscilando entre os números semelhantes aos de Gram-negativo e figuras em torno de 50%. Dentro destes, eles representam problemas de confiabilidade sobre todos os estreptococos do grupo de viridans e não-coagulase não produtor de estafilococos.
Os resultados em casos de fungemia, nos primeiros estudos, foram decepcionantes. Uma extração correta, indispensável neste caso, leva a figuras de identificação acima de 90%, homologada com as obtidas em bacteremias37. O possível impacto de certos componentes de algumas hemoculturas, como o carbono ativo, pode ser levado em conta, o que pode interferir com a identificação. Da mesma forma, um tempo de incubação prolongado da cultura do sangue pode ter um impacto negativo nos espectros obtidos. No entanto, uma vez que o procedimento habitual é realizar MS em, no máximo, algumas horas após a positivização, e mais de 80% de culturas sanguíneas positivas significativas estão nos primeiros 48 h, o impacto clínico desse aspecto não parece transcendente .
Ao avaliar a heterogeneidade das percentagens corretas de identificação nas hemoculturas, deve ser levada em conta que não há critério homogêneo sobre como avaliar os escores obtidos. Alguns autores consideram a identificação correta com valores > 1.7. Outros diminuem essa exigência até 1,5, mas incluem requisitos como a identificação é repetida nas duas ou três primeiras posições da lista de identificação possível, ou que entre as duas primeiras opções oferecidas pela diferença de Maldia-TOF há uma diferença de pontuação de pelo menos 0,3 pontos.
Em geral, não somos a favor de reduzir o nível de requisito na identificação correta nas culturas sanguíneas, uma vez que a existência de uma porcentagem superior de “sem identificações” suporta sempre Um risco menor, do ponto de vista clínico, do que a proliferação de identificações incorretas, o que poderia condicionar os tratamentos inadequados.
Quanto ao uso de métodos de processamento comercial, como sepsityper, os resultados em geral são semelhantes em relação aos métodos de processamento manual. Cada laboratório deve priorizar entre a maior normalização e economia de tempo de processamento que propicia esses métodos, ou economias econômicas (cerca de € 1 / amostra) que envolve o uso de um método manual.
no conjunto, em Maldi- O TOF é um método rápido e confiável para identificação direta de microorganismos em hemoculturas. Sua combinação com métodos que permitem a detecção de mecanismos de resistência a certos antimicrobianos, permite oferecer informações clínicas valiosas a um tempo significativamente menor (24-48 h menos) conforme exigido pela metodologia convencional. Provavelmente, o ponto principal para elucidar, neste momento, é como integrar essa nova atividade em laboratórios de microbiologia, para que as informações oferecidas sejam otimizadas sem supor a sobrecarga sobre os serviços, geralmente não precisamente sobre o técnico de pessoal e com estruturas de tempo muito diversificadas. / p> Identificação direta de outras amostras
O número de estudos relativos à aplicação direta do Mali-TOF em outras amostras biológicas é menor. Como discutido em outra seção, a utilidade de Em Maldia-TOF nessas amostras é determinada por vários fatores: por um lado, a amostra deve vir de uma área estéril sob condições fisiológicas, e em que a infecção, quando aparece, ser geralmente monicrobiano. Por outro lado, tanto a carga bacteriana presente na amostra seja crucial, como o volume de amostra disponível para o estudo. Isso faz com que muitas amostras diferentes de problemas de urina e hemocultura, por baixo volume, geralmente disponíveis (CSF, exsudados purulentos), devido à baixa concentração de microrganismos (LCR, fluido peritoneal em pacientes submetidos à diálise peritoneal) ou pela presença de importantes quantidades de – Proteínas-bacterianas que podem alterar os perfis ou interferir com a ionização de proteínas bacterianas (exsudatos purulentos).
baixa sensibilidade nesses produtos em que a carga bacteriana não é muito alta, e o volume de amostra disponível, Muitas vezes limitada, tornam técnicas moleculares preferíveis para o diagnóstico rápido, mesmo com a limitação de que a gama de microrganismos detectáveis em um único teste é muito menor do que com em Cuzii -tof. Diagnósticos etiológicos de meningite bacteriana ocasionalmente comunicados pela CDRI-TOF no CDR, mas um estudo informado a 21ereccmid indica que, em 183 amostras de CSF, dos quais 14 foram positivos para o método convencional, nenhum foi positivo por Em Cuzid -TOF Sample38, que reafirma o fato de que, do ponto de vista da sensibilidade, os Maldi-Tofs não podem competir com as técnicas moleculares. Em outros tipos de amostra, a experiência é muito limitada, não há protocolos ou dados publicados quanto à sensibilidade e especificidade, e, portanto, o uso clínico de em Maldia-TOF, neste momento, não é pertinente.
Mídia cromogênica
> Desde a aparência do primeiro médio cromogênico (MC), há mais de 30 anos, se tornaram uma ferramenta muito útil para o isolamento diferencial de microorganismos patogênicos. Atualmente, há MC multidão comercializado para a identificação de bactérias e fungos, bem como para o estudo de alguns mecanismos de resistência antimicrobiológicos.
A base desses meios na inclusão de um substrato cromogênico que, sendo hidrolisado por Uma enzima específica presente no microrganismo, dá origem a uma colonação característica de colonação, permite a sua diferenciação.Eles também são também meios seletivos, que inibem o crescimento de outros microrganismos em maior ou extensão, favorecendo a detecção de colônias coloridas. Os principais substratos dos MCS comerciais são derivados indócicos39 que podem ser hidrolisados por galactosidas ou glucosidas, produzindo baixos derivados tóxicos e que não inibem o crescimento bacteriano.
Detecção de microorganismos gram-positivos
Grandes esforços foram dedicados a O desenvolvimento de métodos para a detecção de resistência à meticilina S. aureus (MRSA), dada a sua importância na infecção nosocomial. Nesse contexto, o uso do MC coletou grande importância para o rápido isolamento e identificação dessa bactéria. Existem numerosos MC no mercado para a detecção de MRSA. Um estudo recente40 compara três deles: Cromid MRSA Smart (Smart), geração de MRSA cromado (cromóide) e brilho MRSA (OX2) para triagem de 1.220 amostras de pacientes hospitalizados. A detecção nessas mídias foi corroborada por Em Maldia-TOF, disseminação no ágar com discos de cefoxitina e PCR comercial para Meca e MECC. A sensibilidade às 24 horas foi melhor para o meio inteligente em relação ao meio cromóide, mas não houve diferenças significativas com o ambiente OX2. No entanto, os autores indicam que ainda é necessário, obter resultados ideais, enriquecimento de caldo por 24 h antes da inoculação em qualquer uma das mídias estudadas.
Xu et al.41 avaliações de 5 da mídia mais usada para Detecção de MRSA. Aquele que melhor resultados deste estudo é o Brilliance MRSA 2 (Oxóide Ltd, Termofisher, EUA), com sensibilidade de 65,7% e uma especificidade de 99,8%. O rendimento deste meio melhora se a amostra for inoculada em um caldo de enriquecimento antes de plantá-lo no MC, com uma sensibilidade de 100% e especificidade de 99,1%. A mídia Chromagar MRSA mostra uma sensibilidade de 95% após 24 h de incubação, que atinge 100% se for incubada 48 h. A especificidade em ambos os casos é de 100%. O MULTO BBL O cromo MRSA inclui a cefoxitina em sua composição, o que simplifica a interpretação dos resultados. Este meio foi estudado usando isolados de MRSA obtidos de hemoculturas, com sensibilidade e especificidade de 97,6 e 99%, respectivamente. A Selecção de MRSA foi avaliada utilizando 652 isolada de hemoculturas após a incubação de 18-24 h a 35 ° C, com uma sensibilidade de 99% e especificidade de 98%, que aumentou 99% quando combinado com o teste de coagulase. A cromida média MRSA, que também incorpora a cefoxitina na sua composição, foi estudada em isolados de lesões e culturas sanguíneas. Nas hemoculturas, mostra uma sensibilidade de 97,8% a 24 h, que aumenta 100% a 48 h, e uma especificidade de 99,7% que é mantida (99,6%) a 48 h. Em feridas, sensibilidade e especificidade foram 88,9 e 100% em 24 h e 100% em ambos os casos a 48 h.
O estabelecimento de comparações entre este tipo de mídia de cultura é complexo, uma vez que existem muitos fatores, Além do meio de cultura, o que pode influenciar os resultados (tipo de amostra, concentração de inóculo, tempo de incubação …). No entanto, os dados de sensibilidade e especificidade disponíveis sugerem que qualquer uma das mídias acima mencionadas pode ser usada como uma rotina para a detecção de MRSA com alta confiabilidade.
Outro grupo de bactérias que adquiriram importância no nosocomial da infecção são Enterococci resistente à vancomicina (VRE), ao ponto de que o CDC recomenda o estudo de portadores em altos centros de prevalência. O estudo das transportadoras do VER foi realizado usando o AGAR de Sculin Bilis com azida e vancomicina (Beav). Seu uso envolveu um atraso de diagnóstico de 48 h, além da necessidade de realizar testes complementares para identificação adequada. Para acelerar este processo, vários MCs foram desenvolvidos. Um estudo recente compara cinco mc por vre usando 400 amostras de fezes. Os meios utilizados foram intray Colorx VRE (Biomeddiagnóstico, Cidade Branca, ou), Cromido VRE (Biomérioux, Marcyl’étoile, França), Vreselect (Bio-Rad, Marnes-la-Coquette, França), Hardychrom Vre (Hardy Diagnostics, Santa Maria, CA) e Spectra VRE (Remel, Lenexa, KS), usando Beav Agar e Beav Broth como método de referência. A leitura foi realizada em 24 h. Os resultados da mídia mostra uma sensibilidade entre 89,9 e 94,9%, maior em todos os casos do que os mostrados pelo Agar Beav (84,9%). Os melhores dados de sensibilidade e especificidade continuam a fornecer-lhes o Beav, mas à custa de um atraso de diagnóstico de até 48 h com relação ao MC. O MC mais sensível neste estudo foi cromido VR (94,9%), embora sem diferenças estatisticamente significantes com o resto.
A especificidade e a capacidade de diferenciar espécies de Enterococcus foram semelhantes para os cinco MC. A intrual colorx vr tinha a mais baixa especificidade com 98,8%, enquanto as das outras quatro placas foram de 99,7%. As características de sensibilidade e especificidade desses meios contribuem para a detecção de transportadores da VRE quando isso é pertinente. Eles são, pelo menos, perfeitamente comparáveis ao ágar Beav, diminuindo o tempo de detecção em 24-48 h.
Detecção de microorganismos gramnegativos
em 1979, Kilian e Blow descreve um novo meio de cultura que usa β-glucoronidase como um substrato para detecção direta de E. coli em uroculturas. Estudos de rentabilidade indicaram que este método envolveu uma economia econômica de 46% e uma redução de 64% de tempo de identificação em relação aos métodos convencionais39. Desde então, numerosos MCs foram desenvolvidos com diferentes substratos para a identificação dos principais uopatógenos. Uma das mídias comercializadas recentemente é a Elite CRMID CPS (Biomérieux, Durham, NC) que permite a identificação direta de E. coli, e a identificação presumida de Enterococcus spp., De alguns enterobacteriaceae e as bactérias do grupo Provéea, embora nesses casos A confirmação da identificação é necessária por testes bioquímicos ou em MALDI-TOF.
A eficácia deste meio para a detecção de uroopatogens43 foi avaliada, avaliando o tempo necessário para o diagnóstico e consumíveis utilizados em comparação com o uso de ágar sangue e ágar MacConkey. A concordância deste meio com a metodologia convencional foi de 88% para a urina clinicamente significativa, de 74% para a urina com crescimento não significativo, 69% para contaminados e 95% para placas sem crescimento bacteriano. As principais discrepâncias foram devidas ao crescimento do ágar de sangue, mas não no MC de Bactérias Grampopositivas, em geral, com pouca relevância clínica nas uricações, como Staphylococcus spp. e lactobacillus. Em relação ao tempo decorrido entre a semeadura da amostra e a identificação das colônias, a diferença não foi estatisticamente significativa se os dois meios forem comparados globalmente (27,2 h de média para o método convencional em relação a 26,6h para o MC), mas foi em Aqueles mictórios em que E. coli cresceu em cultura pura (27,1 h no método convencional em comparação com 24,4 h em cromídeo, p
0,0001). Os autores argumentam que este tempo de melhoria é devido à facilidade e confiabilidade da identificação de cor, que evita a necessidade de métodos de identificação complementares.
Media Chromid ™ CPS (CPS4) (BIOMÉRIEUX, ST. Laurent, QC) e Uiselect ™ 4 (URS4) (Bio-Rad, Montreal, QC), foram avaliados44, mais de 903 amostras de urina, comparadas também com o método de ágar de sangue convencional e o Agar MacConkey. A concordância com o método convencional foi de 89,3 e 89,5% para URS4 e CPS4, respectivamente. Se apenas aquelas amostras em que o crescimento foi considerado clinicamente significativo, essa concordância aumentou para 93% no caso de URS4 e 93,1% para o meio CPS4.
Em todos os casos, os autores concordam que a maior vantagem de O uso de MC é a racionalização do processo de identificação, reduzindo a necessidade de testes complementares, o que permite um diagnóstico etiológico mais rapidamente.
Dado o utilitário demonstrado a partir desta ferramenta de diagnóstico, vários grupos estão projetando novo MC , alguns ainda não comercializados, o que pode supor uma alternativa interessante no futuro. Um novo Media45 foi projetado para a detecção de bacteróides, que inclui em sua formulação de 3,4-ciclo-hexanoscuetina-p-d-glicoside, que é alvo de ação de β-glucosidase de bacteroides, e que dá origem à aparência de Colônias de preto. Este meio foi comparado com o ágar Sculinear Bilis em 100 amostras fecais. O MC permitiu a identificação de B. Fragilis em 34 amostras, enquanto em Agar Bilis Esculina, este microrganismo foi detectado em 19 amostras. Além disso, o MC foi muito mais seletivo, já que, em nenhum caso, foi observado crescimento de espécies não-gênero, enquanto em Agar Bilis Esculina observou-se crescimento de microrganismos pertencentes a outros gêneros (Enterobactérias, Clostridium, Candida …) por 34 ocasiões. Os autores também estudam a capacidade desses MCs, complementados com Meropenem e com metronidazol, para detectar a presença de bacteroides resistentes a um ou outro antimicrobiano, demonstrando que em ambos os casos eles são capazes de detectar estirpes resistentes com uma sensibilidade de 100% e especificidade de, pelo menos 85%.
Outro meio inovador desenvolvido é a cromagar enterocolante Yersinia que apresenta uma vantagem sobre o ambiente de cessulodina-irgasan-novabiocina a possibilidade de cepas de crescimento de Y. Enterocolitica serovar O3 e Y. Psedotuberculose, cujo crescimento pode ser visto Inibido no meio CI46.
Detecção de levedura
Embora o primeiro MC tenha desenvolvido para fungos, no início dos anos 90, eles tinham pouco sucesso em sua aplicação clínica por sua capacidade de diferenciação limitada, MC para Mycology desenvolvido no Os últimos 15 anos tiveram um sucesso muito maior, pois permitem uma rápida identificação presumida de diferentes espécies de levedura, um estudo melhor de culturas mistas e a identificação precoce das espécies associadas à resistência antifúngica. Os meios desenvolvidos são baseados, como nos casos anteriores, na presença de substratos para uma ou várias enzimas (β-N-acetil hexossaminidase e, em alguns casos, β-glucosidase ou fosfatase), que permitem uma identificação rápida, pelo menos Por Candida Albicans, e em numerosos casos a identificação definitiva ou presumida de Candida Gabrada, Candida Lusitaniae, Candida Kefyr, C. Parapsilose, Candida Tropicalis e Candida Krusei. Os estudos disponíveis e a experiência já prolongada com este tipo de mídia demonstram uma alta especificidade de especificidade e boa sensibilidade. No entanto, dada a variabilidade de cor e morfologia que pode aparecer em isolados da mesma espécie, a identificação deve ser sempre considerada presumível. Um estudo recente sobre mais de 5.600 amostras clínicas positivas para levedura, mostra que mais de 8% eram culturas mistas, cuja detecção é muito mais provável se o MC for usado, e propõe um algoritmo que combine MC e em Maldia-TOF como procedimento mais apropriado de identificação47.
Conflito de interesses
Os autores declaram não ter nenhum conflito de interesse.