Masse Spettrometria Maldi-TOF. Origini
L’introduzione della spettrometria di massa (MS) Desorbimento laser a matrice-assistito Desorbimento / ionizzazione del volo del volo (Maldi-TOF) è stato, in tutte le probabilità, il cambiamento tecnologico del più grande progetto è avvenuto nella clinica di microbiologia nel ultimo decennio. Tra qualche anno è passato dall’essere una novità promettente, essere una tecnologia completamente integrata nell’attività clinica quotidiana e disponibile, nel nostro paese, nei servizi di microbiologia di numerosi centri ospedalieri1. Per questo è stato necessario per gli spettrometri di massa subire una significativa evoluzione tecnologica. A questo proposito, c’erano due progressi che erano cruciali. Innanzitutto, la progettazione del tempo del sistema di volo (TOF) di W.e. Stephens, nel 1946, che ha permesso la separazione di diverse masse. Quando accelera gli ioni in un campo elettrico e acquisisce tutta la stessa energia cinetica, la velocità acquisita, e quindi il tempo utilizzato per viaggiare il tubo del vuoto, dipende dalla massa della molecola ionizzata, che può essere dedotta dal tempo trascorso questa rotta.
La seconda pietra miliare è stata lo sviluppo di metodi che hanno permesso le proteine intatte ionizzanti che, a causa della loro dimensione, fino ad allora non erano state suscettibili alla ionizzazione, poiché l’alta energia richiedeva per esso ha finito per alterazioni o distruggere la propria proteina. Nel 1987, Köichi Tanaka presenta un nuovo metodo di analisi (desorbimento laser morbido) che ha permesso il trasferimento alle molecole l’energia necessaria per ionizzarli senza rompere i fragili legami chimici. Questa scoperta gli ha conseguito il premio Nobel in chimica nel 2002, insieme a John B. Fenn, per “lo sviluppo di metodi di identificazione e analisi strutturale delle macromolecole biologiche”. Non c’erano più spettrometri di massa basati su questo tipo di analisi, sebbene siano necessarie alcune modifiche nel metodo in modo che l’EM Maldi-TOF sia apparso, in cui il desorbimento degli ioni è favorito da una matrice che assorbe l’energia del laser e parzialmente trasferirlo all’oggetto di studio delle molecole. Anche se queste modifiche hanno migliorato la tecnica, rendendolo più facile e più facile, i responsabili di questi miglioramenti, Michael Karas e Franz Hillenkamp, non sono stati inclusi nel premio Nobel, che ha generato un’importante polemica. Sulla base dei contributi di questi autori ha iniziato a essere possibile, lo studio di macromolecole e biopolimeri da parte della SM, che ha aperto nuovi campi di applicazione per questa tecnologia.
nei suoi quasi 30 anni di vita, Maldi Em – TOF è stato utilizzato per l’analisi quantitativa e qualitativa delle proteine di varie origini. Inizialmente, è stato applicato a proteine precedentemente isolate o piccole serie di proteine, ma grazie ai progressi tecnici sia nella strumentazione che negli strumenti per l’analisi del computer dei dati, lo studio di grandi gruppi di proteine può ora essere affrontato.
La possibilità di iniziare a studiare proteine complesse e non solo peptidi piccoli, ampliato notevolmente i potenziali campi di applicazione della SM. Uno di questi è stato l’identificazione dei microrganismi che, poiché la procedura è stata semplificata e il software necessario è stato migliorato per sfruttare i dati grezzi forniti dallo spettrometro, derivati rapidamente nella loro applicazione clinica.
massa sptaltometrica Malzi-TOF in microbiologia clinica
Fino all’introduzione di EM Maldi-TOF, l’identificazione batterica, anche con progressi significativi come la creazione di gallerie di identificazione miniaturizzata e l’automazione della sua inoculazione e lettura, ha continuato a essere nutrito dai metodi sviluppati da batteriologia classica La pratica dei sistemi di identificazione ha continuato ad essere basato sulla fermentazione degli zuccheri e del loro rilevamento attraverso il cambio di pH generato, la metabolizzazione di altri substrati e la produzione di diversi metaboliti e attività enzimatiche rilevabili da metodi chimici. Tutti questi metodi hanno adoleto diverse limitazioni:
- •
crescita batterica richiesta, che supponeva, nella maggior parte dei casi, un periodo di incubazione di almeno 16-18 ore dalla sua inoculazione fino alla lettura.
- •
ha mostrato problemi di identificazione in tutti quei microrganismi con difficoltà a crescere nei media liquidi utilizzati per l’inoculazione di questi pannelli, nonché nei microrganismi con bassa attività biochimica ed enzimatica.
- •
È stato necessario considerare il margine di errore derivato dal fatto che, gli individui della stessa specie, possono avere comportamenti diversi contro vari substrati.
Queste limitazioni, sebbene fossero conosciute e assunte, sono diventate più chiaramente brevetti quando, data le discrepanze osservate in alcuni studi tra MSZ-TOF e identificazione convenzionale, Il sequenziamento del RNR 16S ha dimostrato che, nella stragrande maggioranza dei casi, la corretta identificazione corrispondeva a quella fornita dallo EM MALDI-TOF2.
A tale riguardo, l’EM Maldi-TOF ha ovvi vantaggi:
- •
indipendentemente dalla possibilità di identificare i microrganismi direttamente da alcuni campioni, che viene trattata in un’altra sezione, anche la crescita del piatto molto scarsa o precoce consente un’identificazione affidabile in un breve periodo di tempo , risparmiando così, almeno, quelle 16-18 h di crescita nei sistemi di identificazione biochimica.
- •
L’analisi del profilo proteico del microrganismo nello spettro del 2- 20kd, che è dove la maggior parte delle proteine delle costine sono posizionate Osomic, offre la stragrande maggioranza delle specie batteriche un profilo specifico, che consente la differenziazione dal resto con un’affidabilità simile a quella offerta dal sequenziamento del RNR 16s.
A Questo deve essere aggiunto che, l’introduzione di questa tecnologia, ha notevolmente ampliato la gamma di generi e specie che siamo in grado di identificare con affidabilità, con un metodo in grado di essere utilizzato in routine. Ciò ha persino portato ad una rivalutazione del ruolo dei patogeni dei microrganismi che, a causa della difficoltà della loro identificazione da parte dei metodi classici, erano molto probabilmente infradiagnosid3.4.
Già nel 1975, Anhalt e FenselAU propongono l’uso della SM per l’identificazione dei microrganismi5. Vent’anni dopo viene pubblicato il primo studio che dimostra l’efficacia del Maldi-TOF EMS per l’identificazione dei microrganismi da parte di cellule piene6.
Nel 2009 è stato pubblicato che probabilmente era un articolo chiave da dare a sapere in generale , Specialisti coinvolti nella diagnosi di malattie infettive, le possibilità di EM Maldi-TOF7. Studia più di 1600 isolati che includono batteri grampostitivi e gramnegativi, aerobici e anaerobici, ottenendo il 95,4% delle identificazioni corrette. Questo articolo è rivolto a questo articolo che è stato poi trasceso per l’utilità pratica di questa metodologia: la disponibilità di banche dati di microrganismi sufficientemente ampi, sia dal punto di vista qualitativo (numero di generi e specie incluse) e quantitativi (gli autori dimostrano che la corretta probabilità di identificazione è maggiore per quei microrganismi che esistono almeno dieci profili diversi introdotti nel database).
Da questo momento, l’espansione rapida dell’uso si verifica di questa tecnologia in microbiologia clinica. La prima pubblicazione in Spagna avviene nel 2010, mostrando una correlazione con la metodologia convenzionale, a livello di specie, 100% in gramsitivo e 87,7% in gramnegative8. Da allora, il numero di pubblicazioni relative a diversi aspetti dell’uso clinico di EM Maldi-TOF, ma soprattutto all’identificazione dei microrganismi, è stato esponenziale7-12.
Identificazione dei batteri gramnegativi usando Maldi-TOF
Gli studi dimostrano che l’efficienza nell’identificazione degli entribatteri e di altri gram-negative è eccellente, compresi sia i gram-negativi più comuni in clinicamente e altri meno frequenti o più complicati da identificare con la metodologia classica (diverse specie di Yersinia, Aeromonas, Plasiomonas ,. Brucella, Francisella, Achromobacter, Stenotrophomonas, Burkholderia …). Come in altri casi, i problemi di identificazione sono stati quasi sempre collegati a insufficienze di banche dati anziché limitazioni del metodo, come si è verificato in alcuni studi con generi come Ralstonia, Elizabethkingia o Sphyngobacterium13. Inizialmente, sono state sollevate alcune limitazioni che potrebbero avere una maggiore trascendenza clinica, come l’incapacità di distinguere tra Escherichia Coli e Shigella14 o la difficoltà di distinguere i sierovarre di Salmonella Enterica. Studi più recenti suggeriscono che è possibile salvare queste limitazioni. Utilizzo dei programmi per computer come Flexanalysis e ClinproTools (Bruker Daltonica GmbH, Germania), sono stati identificati picchi specifici che consentono diversi da E. Coli e Shigella nel 90% dei casi15. Altri autori hanno dimostrato che questa differenziazione è possibile anche senza utilizzare software aggiuntivo, semplicemente aumentando il numero e la varietà di profili di E. Coli e Shigella presente nel database di riferimento16. Con questo metodo, gli autori identificano correttamente 60/64 isolati di E. Coli e 110/116 isolati di Shigella.
A tutti coloro che hanno usato l’EM Maldi-TOF per l’identificazione, è consisteva che, con la solita metodologia, l’identificazione della salmonella a livello di genere è affidabile, ma oltre il livello di genere è molto Di meno. Tuttavia, ci sono già pubblicazioni 2004 che suggeriscono l’esistenza di picchi specifici che potrebbero consentire l’identificazione dei sierovarre con maggiore affidabilità17. Uno studio recente suggerisce l’esistenza di picchi che consentono di identificare in modo affidabile Typhi18 Serovar. Poiché gli elementi differenziali sembrano essere esistenti, un’estensione del database di riferimento con un numero maggiore di altoparlanti, almeno, i serovars più frequenti, probabilmente migliorerebbero la risoluzione in questo caso particolare.
in altri casi, Come è la differenziazione tra le specie del complesso Enterobacter Cloacaee, la capacità dell’em Maldi-TOF è di circa l’80%, che senza essere ottimale, rappresenta un miglioramento sensibile rispetto alla metodologia convenzionale19.
sul Altra parte, come già accennato, la corretta identificazione dei microrganismi precedentemente identificati in modo errato, come il genere Raulatella, frequentemente identificato da metodi classici come la Klebsiella o come enterobacter, consentono una visione più reale del suo ruolo di patogeni, precedentemente sottovalutato3.
Identificazione dei batteri di grampositivas con Maldi-TOF
L’identificazione di microrganismi gram-positivi di solito mostra figure in qualche modo inferiori, dovute In alcuni casi alla difficoltà di sdraiarsi sul muro, e in altri alla somiglianza tra specie. In generale, il metodo diretto (applicazione della colonia insieme alla matrice sulla piastra dello spettrometro) è solitamente sufficiente per ottenere una buona identificazione dalla colonia, ma a volte la precedente estrazione con etanolo / acido formico può essere necessario per ottenere valori di identificazione ottimali. Insieme, offre risultati eccellenti nell’identificazione di Staphylococcus Aureus e altre specie e sottospecie di stafilococchi, produttori e non produttori di coagulasi, nonché da enterococchi e altri morti meno frequenti come micrococcus, Gemella, Rothia, ecc. Ha anche dimostrato la sua utilità nell’identificazione dei bacilli gram-positivi, compresa una complessa identificazione dei metodi classici (Listeria, Lactobacillus, Corynebacterium, Nocardia, Actinotignum …) 20.
Inoltre, come in Il caso dei gramnegativi, l’uso sistematico di questa tecnologia sta cambiando considerazione come patogeni di alcuni microrganismi, come lo staphylococcus lugdunensis21. Insieme, i dati disponibili lo rendono consigliabile, nel caso di Staphylococci da utilizzare, come in qualsiasi altro caso, i database che offrono un numero maggiore di specie e un numero ragionevolmente elevato di profili per specie e utilizzare un metodo di estrazione al posto di Applicazione diretta, nonché applicare il microrganismo sulla piastra in duplicazione. La riduzione del punteggio richiesto per convalidare un’identificazione a livello di specie ai valori > 1.7, invece di > 2.0 come è comune , poiché alcuni autori22 propongono, possono aumentare il numero di identificazioni a livello di specie, ma in cambio di una diminuzione della specificità la cui rilevanza può essere discutibile.
Probabilmente il gruppo di Streptococchi è quello che presenta maggiori problemi tra gramsivives. Mentre alcune delle principali specie patogeniche (Streptococcus Pyogenes, Streptococco Agalactiae) sono identificate con alta affidabilità, Streptococcus Pneumoniae (S. Pneumoniae) è sempre stata fonte di problemi, a causa della sua confusione con altre specie del Gruppo Mitis. Le squadre principali in uso nel nostro paese identificano S. Pneumoniae con affidabilità. L’errore più comune è l’identificazione errata di altri streptococchi del gruppo Mitis come S. Pneumoniae, in particolare con il database di Bruker23. Alcune picchi sono stati descritti come S. Pneumoniae, quindi alcuni autori raccomandano il loro uso per confermare queste identificazioni23. Tuttavia, l’ultima libreria di riferimento di Bruker Dytonics (Libreria MBT 6903 MSP) continua a raccomandare l’uso di altri test (Optoquina, solubilità in Bile) per differenziarli.
Anche all’interno dello stesso gruppo, la qualità di L’identificazione delle diverse specie può variare considerevolmente, come avviene all’interno del Gruppo Anginosus Streptococcus.
In ogni caso, come con Staphylococci, l’uso di un database così ampio e completo possibile e l’identificazione in duplicazione E con i protocolli che includono l’estrazione sono consigliati per ottimizzare i risultati (figura 1).
Grampositive, gram-negativo, anaerobi e lievito che lavora utilizzando Maldi-TOF.
Identificazione dei batteri Anaerobias attraverso Maldi-TOF
L’identificazione dei batteri anaerobici, sia grambous che gramnegativi, ha un alto grado di affidabilità, identificando correttamente le specie più comuni (batteroid, precipizio, porfiomonas, fusobacterium, clstridium, actinomyces …). L’applicazione del Maldi-TOF EM in questo campo ha anche dimostrato che l’identificazione biochimica è stata molto meno affidabile di quanto fosse pensato3. La combinazione di semplicità, rapidità e affidabilità rendono l’EM Maldi-TOF nel metodo di scelta per l’identificazione di routine dei batteri anaerobici in clinica. L’estrazione o meno il campione non sembra modificare significativamente i risultati, né fa il mezzo di coltura di origine. Un fattore decisivo, come in altri casi, è l’ampiezza dei database. Già uno dei primi studi condotti in Spagna3 ha dimostrato che la maggior parte degli errori nell’identificazione dei batteri anaerobici è dovuta all’assenza di profili di riferimento delle specie corrispondenti nel database. Gli studi più anziani offrono un’efficacia nell’identificazione del 75-80%, mentre studi più recenti, con database più completi o database specifici offrono figure superiori al 90%. La capacità di identificare i ribotipi difficilari di Clostridium, suggeriti da alcuni autori, non è stato dimostrato definitivamente.
Identificazione del lievito e dei funghi filamentosi attraverso Maldi-TOF
L’identificazione del lievito è stata mostrata dagli inizi eccellenti risultati, differenziando anche Specie complicate di discriminazioni da metodi convenzionali, come il complesso di parasilosis candida. Tuttavia, i risultati in relazione ai funghi filamentosi sono stati molto più irregolari. Alcuni studi iniziali hanno mostrato buoni risultati che studiano selettivamente spore, ma questo metodo non è impraticabile in un laboratorio clinico, quindi la maggior parte degli studi sono stati indirizzati allo studio congiunto di spore e ife. Nel caso di funghi, l’estrazione convenzionale con acido formico e acetonitrile migliora significativamente i risultati. Altri metodi che sono stati testati, come l’uso di perline di vetro per frammentare le pareti, non sembrano offrire risultati significativamente superiori all’estrazione convenzionale.
Un problema posto da funghi filamentosi è l’esistenza di differenze significative Nei profili proteici ottenuti in base all’antichità delle colture e persino tra diverse sottoculture dello stesso CEPA24. La soluzione a questo attraversa l’elaborazione di database più ampi e più complessi, che incorporano i profili da un numero maggiore di ceppi e colture di diversa antichità. Alcuni produttori raccomandano la rimozione dopo un raccolto di un giorno in brodo, sebbene ciò abbia un impatto su un ritardo di 24 ore nell’emissione del risultato. Uno studio condotto nel 201125 mostra che un database ben elaborato e sufficientemente complesso è fondamentale e che può migliorare l’identificazione di funghi filamentosi alle figure simili a quelle ottenute con batteri e lieviti. Sfortunatamente, l’elaborazione e la convalida di questi database sono complesse e non sono disponibili per molti utenti e sono disponibili pochi sviluppati. Pertanto, e anche per scopi di standardizzazione, è probabilmente più appropriato utilizzare i database dei produttori, che devono essere comodamente ampliati.
Attualmente Bruker Dytonics, il cui database di funghi filamenti, nella sua versione MBT 6903 MSP Library , Comprende 25 generi e 42 specie, raccomanda la cultura in media pernottamento liquido, seguita da centrifugazione, lavaggio ed estrazione con etanolo, acido formico e acetonitrile (figura 2). MS-Vitek, nella sua versione 3.0, include 32 Genera e 81 specie e SARAMIS nella sua versione RUO 4.13, include un database più ampio, con 45 generi e 168 specie. Biomérieux non raccomanda la cultura in mezzo liquido, ma un’estrazione convenzionale con etanolo, acido formico e acetonitrile (figura 2). Nonostante tutto, uno studio recente che utilizza la metodologia raccomandata di Bruker identifica correttamente, a livello di specie, solo il 72% degli Isolando26 e uno studio molto recente con Vitek 3.0 identifica correttamente il 66,8% di 318 isolato, in particolare per le carenze del database27. Pertanto oggi, l’identificazione di funghi filamentosi da EM Maldi-TOF non può ancora sostituire completamente la metodologia di identificazione convenzionale.
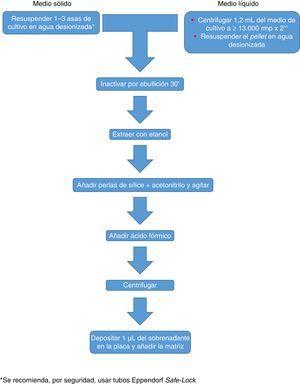
Elaborazione mycobatterica consigliata per il sistema Biotiyper Maldi.
div id = “4895790f6a”>
Identificazione dei mycobatteri usando Maldi-TOF
Le limitazioni della metodologia tradizionale per l’identificazione dei micobatteri, su tutto in cui si riferisce al tempo di risposta, loro Ho fatto la totalità pratica dei laboratori ha decantato dai metodi di identificazione molecolare. L’uso di EM Maldi-TOF per l’identificazione di Mycobacteria è un’alternativa altrettanto veloce e più economica delle tecniche molecolari, ma mostra peculiarità in diversi aspetti. Da un lato, i metodi diretti di analisi o che implicano un’elevata concentrazione o manipolazione dei microrganismi precedentemente alla sua inattivazione, non sono raccomandati da una questione di sicurezza biologica. L’uso di un metodo di estrazione che garantisce la rottura delle cellule migliora la qualità degli spettri e aumenta la sicurezza della procedura.
Diversi fattori possono influenzare l’efficacia dell’identificazione dei micobatteri di Em Maldi – TOF. Come è stato anche mostrato con funghi, i mycobatteri possono avere profili proteici molto diversi a seconda dell’antichità delle colture, quindi è importante che la diagnosi includa profili proteici delle colture di diversa antichità per ogni microrganismo. È stato anche parlato della possibile formazione di polimeri che mascherano le dimensioni effettive delle proteine caratteristiche utilizzate per l’identificazione. Tutto ciò rende l’identificazione dei mycobatteri una procedura meno prevedibile in termini dei suoi risultati rispetto a quella di altri batteri, come dimostra l’eterogeneità dei dati pubblicati. Negli ultimi aggiornamenti, le biblioteche hanno avuto miglioramenti significativi. Pertanto, ad esempio, la biblioteca melobacticabile Biotyyper include 149 specie e la versione 3.0 di Vitek MS incorpora 48 nuove specie rispetto alla versione precedente.
La procedura inizialmente raccomandata da Bruker ha incluso una serie di passaggi (adeguamento a Una certa densità ottica, diverse fasi di centrifugazione e risospensione) che ha assunto un rischio biologico probabilmente inutile, quindi sono stati sostituiti da un singolo passo di trattamento con il calore. Il metodo raccomandato attualmente include diversi passi di riscaldamento e lavaggio, seguiti da un trattamento con palline di silice e acetonitrile. Successivamente, vengono trattati con acido formico, applicato sul piatto e coperto con la matrice (figura 3). Al contrario, il metodo sostenuto dalla parte Biomérieux di un trattamento iniziale con 70% di etanolo e palline di silice, dopo di che il campione viene estratto con acido formico e acetonitrile, applicato sulla piastra e coperto con la matrice (figura 4).
Elaborazione mycobatterica consigliata per il sistema VITEK MS.
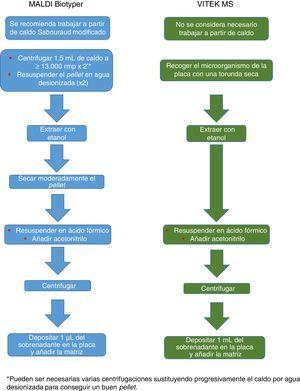
Elaborazione fungine Consigliato per Maldi Biotiyper e Vitek MS Systems.
Uno studio comparativo tra entrambi i metodi effettuati di recente NTE28 mostra che, sebbene l’identificazione tenda ad essere qualcosa di meglio a partire da metà 7h10 rispetto a Lowenstein Jensen, le differenze sono piccole in entrambi i sistemi. Tuttavia, dimostra che il metodo di elaborazione del campione è fondamentale, e non è intercambiabile tra i sistemi. I campioni ottenuti con il metodo di estrazione Bruper forniscono risultati migliori nel loro spettrometro e il trasformato con il metodo Vitek lo fanno con Salamis e Vitek MS stesso, anche se la differenza in questo caso è inferiore. Insieme, Vitek MS sembra essere meno influenzato dal metodo di estrazione. Per quanto riguarda l’efficienza nell’identificazione, Bruker Biotiyper ha identificato un punteggio ≥1,8 81,5% degli isolati, mentre SARAMIS e VITEK MS 3.0 identificati con un’affidabilità ≥90% di 85,4 e rispettivamente 89,2%. Usando questi stessi criteri, il 22,6% degli isolati identificati da Bruker Biotiyper doveva essere estratta su più di un’occasione per raggiungere l’identificazione, che si è verificata del 21,5% con Saramis e del 14,2% con VITEK MS 3.0.
In relazione ad altri microrganismi con struttura parietale vicino a quello dei micobatteri, per il tempo è stato considerato che la sua identificazione era problematica a causa della composizione del suo muro. Tuttavia, è stato dimostrato che, se un database è disponibile con riferimenti sufficienti, un metodo di estrazione simile a quello utilizzato con i mycobatteri può offrire buoni risultati29.
Tutti i dati riferiti sopra suggeriscono che lo spettro dei microrganismi suscettibili L’identificazione di EM Maldi-TOF è straordinariamente ampia e che, con contabilizzazione delle eccezioni, il fattore limitante della capacità del Maldi-TOF emette quasi sempre più nella completezza dei database di riferimento rispetto alla capacità del metodo per ottenere profili affidabili di quasi tutti i microrganismi batterici in grado di crescere nei media di coltura. In effetti, le aziende di produzione sembrano essere consapevoli del problema; Pertanto, l’ultima biblioteca di riferimento pubblicata da Bruker Daltonica (BBT 6903 MSP Library) incorpora 938 nuovi profili, di cui il 17,2% mirano a coprire nuovi generi e specie (15 nuovi generi e 96 nuove specie specificamente), in particolare anaerobes e funghi, Ma l’82,8% mira a crescere la diversità dei profili all’interno delle specie già contemplate nelle precedenti biblioteche. Allo stesso modo, Vitek MS versione 3.0 include 242 nuove specie batteriche, tra cui 48 nuovi melobatteri e 15 notti, e 55 nuovi funghi, con una media globale di oltre 10 specie isolati e 25 specie per specie.
Identificazione dei microrganismi, di Maldi-TOF, dal campione diretto
la possibilità di identificazione dei microrganismi prima della crescita delle colonie è stata, fin dall’inizio, un obiettivo molto attraente, quindi suppone il tempo di essere in grado di stabilire un Trattamento empirico più orientato, ridurre l’emergere della resistenza e ottimizzare la spesa30. L’EM Maldi TOF, applicato al campione diretto, è diventato uno strumento molto utile in gravi infezioni con alto tasso di morbilità come batteriemia e fungemie, sebbene il lavoro diretto sui campioni abbia inconvenienti che possono limitare l’utilità della tecnica. Nei campioni con bassa densità microbica non è possibile eseguire un’identificazione corretta, poiché la quantità di proteine batteriche presenti è insufficiente. Qualcosa di simile si verifica con i campioni polimmetici, poiché può essere generato uno spettro proteico aberrante, come risultato della miscela di diversi profili, o ignorando direttamente il microrganismo che è in una proporzione minore. Alcuni miglioramenti del software sono stati sviluppati, che a volte possono rilevare, e anche identificare i microrganismi coinvolti separatamente, ma richiede attentamente studiati e probabilmente raffinati ad avere una vera utilità. Nella maggior parte dei metodi che funzionano direttamente sul campione, e prima della possibile presenza di microrganismi intracellulari, è consigliabile utilizzare i metodi che includono la lisi cellulare.
Non tutti i campioni sono validi per questo tipo di studio. Il campione ideale è quello che proviene da un’area abitualmente sterile, che può ospitare elevate concentrazioni di microrganismo e in cui non ci sono limitazioni significative relative al volume del campione. Campioni da aree abitualmente colonizzate (pelle, sgabello, apparato respiratorio ad alta …) genereranno profili aberranti in tutte le probabilità. Un problema tecnico da tenere in considerazione quando si lavora direttamente sul campione è il volume del campione disponibile, che in casi come urina o culture del sangue non è di solito un problema, ma in altri con liquido cerebrospinale può essere, e che sarà come Molto più importante come minore è la concentrazione batterica. Inoltre, i campioni con alto contenuto proteico della fonte alieno al microrganismo possono anche generare problemi al momento della sua interpretazione31.
Tuttavia, il vantaggio che è, specialmente nelle emoculture, sorpassa 24 ore Il microrganismo e orientamento del trattamento in particolare, ha effettuato l’uso diretto della SM in campione, in particolare in questi casi, ha diffuso.
Identificazione diretta dai campioni di urina
L’urina è uno dei campioni che meglio si adattano a Condizioni ideali per lavorare con EM Maldi-TOF sul campione diretto. È un fluido sterile in condizioni fisiologiche, il carico batterico nell’urina infetta è elevata nella maggior parte dei casi, e le limitazioni riguardanti il volume del campione e ottenendo nuovi campioni, se necessario, sono minimi.
Studi con eccellente I risultati sono stati pubblicati, con concordanza nell’identificazione che raggiunge il 90-95% 32.33 rispetto ai sistemi automatizzati.Sono stati descritti diversi protocolli di elaborazione dei campioni, tutti coincidono nella necessità di una pre-screening (citometria del flusso, colorazione grammo) per discriminare tra campioni positivi e negativi e limitare l’uso di SM per i campioni inizialmente positivi34.
Con piccole varianti, la preparazione dei campioni di solito consiste nell’inserirle a una serie di centrifugazioni e lavata con acqua deionizzata, quindi eseguire la procedura convenzionale del Maldi-TOF. Le modifiche sono state apportate incorporando l’aliquota campione SDS al 10% 33 per migliorare il rilascio di proteine, o Tween-8034 per migliorare i risultati. I risultati ottenuti sono in genere migliori con Gramnegative che con grampostività e funghi, molto migliorando l’affidabilità quando i conteggi sono alti, sopra 105FC / ml.
Un disagio di questo metodo è stato la necessità di eseguire il Studio di sensibilità attraverso la metodologia convenzionale. Un recente studio suggerisce che, una volta che l’identificazione di EM Maldi-TOF, lo stesso sedimento può essere utilizzato per eseguire un disco di dischi antibiogramma con risultati eccellenti, che consente di ridurre in 24 ore lo studio completo35. L’importante cambiamento organizzativo che l’introduzione di questa metodologia suppone e le infezioni in cui, spesso, sono state diagnosticate alcune ore non sono critiche per la gestione del paziente, hanno reso la diagnosi di infezione urinaria attraverso EM Maldi-TOF non ha avuto il Penetrazione ha avuto, ad esempio, nel caso delle culture del sangue. È necessario raggiungere una maggiore sensibilità e una maggiore normalizzazione dei metodi per considerare la loro introduzione nella routine clinica.
Identificazione diretta dalle emoculture
L’istituzione del corretto trattamento empirico è decisivo nell’evoluzione del batteriemia La probabilità che il trattamento stabilito sia corretto, sarà maggiore più accurato e concreto le informazioni che siamo in grado di fornire rapidamente. In questo aspetto, la possibilità offerta dallo EM Maldi-TOF di ottenere un’identificazione affidabile in un breve periodo di tempo dopo la positivizzazione della cultura del sangue è un progresso evidente.
numerosi protocolli di elaborazione di sangue-culturali hanno stato descritto (centrifugazione differenziale, lisi cellulare ed estrazione di proteine con metodi chimici, separazione del gel …). La maggior parte di loro si basa sulla lisi e sull’eliminazione dei componenti cellulari, sia da centrifugazioni che da lavaggi, in cui diversi composti vengono utilizzati come saponina, cloruro di ammonio o SDS o utilizzando tubi con gel separatore siero e attivatore della coagulazione. In breve, ciò che viene cercato con questi metodi è isolare e concentrare i microrganismi fino a raggiungere almeno 105-107FC / ml, concentrazione a cui la quantità di proteine è sufficiente per generare profili adeguati in EM Maldi-TOF.
Nel caso di emocultures c’è una procedura commerciale approvata (Sepsityper, Bruker Daltoniks GmbH, Germania), costituita dall’aggiunta di una soluzione di lisi a un’aliquota da 1 ml della coltura del sangue, dopodiché il campione è soggetto a diversi passaggi del lavaggio E centrifugazione e, infine, ad un’estrazione convenzionale con etanolo e acido formico36.
I risultati del trattamento diretto delle culture del sangue sono generalmente buoni, con corrette percentuali di identificazione dell’80-90%, anche se con alcune sfumature. L’identificazione dei gram-negativi è solitamente corretta nel 90-95% dei casi, mentre nei gramsivive è molto più eterogeneo, oscillante tra figure simili a quelle del gram-negativo e dei figure intorno al 50%. All’interno di questi, rappresentano problemi di affidabilità rispetto a tutti gli streptococchi del gruppo Viridans e non producono i non produttori non coagulasi.
I risultati in casi di fungemia, nei primi studi, sono stati deludenti. Una corretta estrazione, indispensabile in questo caso, porta a figure di identificazione superiore al 90%, omologate con quelle ottenute in battercallemias37. Il possibile impatto di determinati componenti di alcune emoculture, come il carbonio attivo, può essere preso in considerazione, che può interferire con l’identificazione. Allo stesso modo, un tempo prolungato di incubazione della cultura del sangue può avere un impatto negativo sugli spettri ottenuti. Tuttavia, poiché la consueta procedura è quella di eseguire la SM in, al massimo, alcune ore dopo la positivizzazione, e oltre l’80% delle significative culture del sangue positivo sono nelle prime 48 ore, l’impatto clinico di questo aspetto non sembra trascendente .
Nel valutare l’eterogeneità delle percentuali di identificazione corretta nelle emoculture, deve essere presa in considerazione che non vi è alcun criterio omogeneo in merito a come valutare i punteggi ottenuti. Alcuni autori considerano l’identificazione corretta con i valori > 1.7. Altri diminuiscono questo requisito fino a 1,5, ma includono requisiti come l’identificazione è ripetuta nelle due o tre le prime posizioni del possibile elenco di identificazione, o che tra le prime due opzioni offerte dalla differenza di Maldi-TOF c’è una differenza di punteggio di Almeno 0,3 punti.
In generale, non siamo a favore della riduzione del livello di requisito nella corretta identificazione nelle culture del sangue, poiché l’esistenza di una percentuale più elevata di “Nessuna identificazione” supponga sempre un rischio inferiore, dal punto di vista clinico, rispetto alla proliferazione di identificazioni errate, che potrebbero condizionare i trattamenti inadeguati.
per quanto riguarda l’uso dei metodi di elaborazione commerciali, come sepsityper, i risultati in generale sono simili in relazione ai metodi di lavorazione manuale. Ogni laboratorio deve dare la priorità tra la maggiore normalizzazione e il risparmio di tempo di elaborazione che propiziano questi metodi o risparmi economici (circa € 1 / campione) che comporta l’uso di un metodo manuale.
In Set, Em Maldi- TOF è un metodo veloce e affidabile per l’identificazione diretta dei microrganismi nelle emoculture. La sua combinazione con metodi che consentono di rilevare meccanismi di resistenza a determinati antimicrobici, consente di offrire preziose informazioni cliniche in un tempo significativamente inferiore (24-48 ore meno) come richiesto dalla metodologia convenzionale. Probabilmente il punto principale per ilucidato, in questo momento, è come integrare questa nuova attività nei laboratori di microbiologia, in modo che le informazioni offerte siano ottimizzate senza supporre un sovraccarico sui servizi, di solito non esattamente sopra il tecnico del personale e con strutture temporali molto diverse.
Identificazione diretta da altri campioni
Il numero di studi relativi all’applicazione diretta del Mali-TOF ad altri campioni biologici è più piccolo. Come discusso in un’altra sezione, l’utilità di Em Maldi-TOF in questi campioni è determinata da diversi fattori: da un lato, il campione deve provenire da un’area sterile in condizioni fisiologiche e in cui l’infezione, quando appare, memorizzare Di solito essere monomicrobico. D’altra parte, sia il carico batterico presente nel campione sono cruciali, come il volume del campione disponibile per lo studio. Ciò rende molti diversi campioni di problemi di urina ed emocultura, di solito, per basso volume di volume, solitamente disponibili (CSF, purulento essudati), a causa della bassa concentrazione di microrganismi (CSF, fluido peritoneale nei pazienti sottoposti a dialisi peritoneale) o per presenza di importanti quantità di quantità -Segni proteine in grado di modificare i profili o interferire con la ionizzazione delle proteine batteriche (essudati purulenti).
bassa sensibilità in quei prodotti in cui il carico batterico non è molto alto e il volume del campione disponibile, Spesso limitato, rendono le tecniche molecolari preferibili per una diagnosi rapida, anche con la limitazione che la gamma di microrganismi rilevabili in un singolo test è molto più basso rispetto a EM Cuzii -Tof. Le diagnosi eziologiche di meningite batterica hanno occasionalmente comunicato da CDri-TOF sul QCSF, ma uno studio riportato a 21ereccmid ha indicato che, su 183 campioni di SCSF, di cui 14 sono stati positivi per il metodo convenzionale, nessuno è stato positivo da EM Cuzid -Tof diretto Sample38, che ribadisce il fatto che, dal punto di vista della sensibilità, il Maldi-TOF non può competere con le tecniche molecolari. In altri tipi di campione, l’esperienza è molto limitata, non ci sono protocolli o dati pubblicati sulla sensibilità e specificità, e quindi l’uso clinico di EM Maldi-TOF, in questo momento, non è pertinente.
Media cromogeni
Poiché l’aspetto del primo mezzo cromogenico (MC), più di 30 anni fa, sono diventati uno strumento molto utile per l’isolamento differenziale di microrganismi patogeni. Ci sono attualmente la folla MC commercializzata per l’identificazione di batteri e funghi, nonché per lo studio di alcuni meccanismi di resistenza antimicrobica.
la fondazione di questi mezzi nell’inclusione di un substrato cromogenico che, essendo idrolizzato da Un enzima specifico presente nel microrganismo, dà origine a una caratteristica colonia di colonizzazione, consente la sua differenziazione.Sono anche anche mezzi selettivi, che inibiscono la crescita di altri microrganismi a una maggiore o misura, favorendo il rilevamento delle colonie colorate. I principali substrati degli MC commerciali sono i derivati indtionlicici39 che possono essere idrolizzati da galactosidas o glucosidasi, producendo bassi derivati tossici e che non inibiscono la crescita batterica.
Rilevamento di microrganismi gram-positivi
Grandi sforzi sono stati dedicati Lo sviluppo di metodi per il rilevamento del resistente alla meticalina S. Aureus (MRSA), data la sua importanza in infezione nosocomiale. In questo contesto, l’uso di MC ha raccolto grande importanza per il rapido isolamento e identificazione di questo batterio. Ci sono numerosi mc nel mercato per il rilevamento dell’RSA. Un recente studio40 confronta tre di loro: Chromid MRSA Smart (Smart), Chromid MRSA Fist Generation (Chromid) e Brilliance MRSA (OX2) per lo screening di 1.220 campioni di pazienti ospedalizzati. Il rilevamento in questi media è stato corroborato da EM Maldi-TOF, la diffusione su Agar con dischi cefoxitina e PCR commerciale per la Mecca e il MECC. La sensibilità a 24 ore era migliore per il mezzo intelligente rispetto al mezzo cromidico, ma non c’erano differenze significative con l’ambiente OX2. Tuttavia, gli autori indicano che è ancora necessario, per ottenere risultati ottimali, arricchimento del brodo per 24 ore prima dell’inoculazione in uno qualsiasi dei media studiati.
xu et al.41 recensioni di 5 dei media più usati per Rilevamento MRSA. Quello che meglio risulta in questo studio è la brillantezza MRSA 2 (OXOD LTD, Termofisher, USA), con una sensibilità del 65,7% e una specificità del 99,8%. La resa di questo mezzo migliora se il campione è inoculato in un brodo di arricchimento prima di piantarla nell’MC, con una sensibilità del 100% e una specificità del 99,1%. Il media Chromagar MRSA mostra una sensibilità del 95% dopo 24 ore di incubazione, che raggiunge il 100% se è incubato 48 ore. La specificità in entrambi i casi è del 100%. Il media BBL Chrome Agar MRSA comprende cefoxitina nella sua composizione, che semplifica l’interpretazione dei risultati. Questo mezzo è stato studiato utilizzando isolati MRSA ottenuti da emoculture, con una sensibilità e specificità rispettivamente del 97,6 e del 99%. La selezione MRSA è stata valutata utilizzando 652 isolati dalle emoculture dopo 18-24 h incubazione a 35 ° C, con una sensibilità del 99% e una specificità del 98%, che è aumentata del 99% quando è combinato con il test della coagulase. Il Medio Chromid MRSA, che incorpora anche il cefossitino nella sua composizione, è stato studiato in isolati da feriti e culture del sangue. Nelle emocultures mostra una sensibilità del 97,8% a 24 ore, che aumenta il 100% a 48 ore e una specificità del 99,7% mantenuta (99,6%) a 48 ore. Nelle ferite, la sensibilità e la specificità erano 88,9 e 100% a 24 ore al 100% in entrambi i casi a 48 ore.
L’istituzione di confronti tra questo tipo di media dei media è complessa, poiché ci sono molti fattori, A parte il mezzo di cultura, che può influenzare i risultati (tipo di campione, concentrazione di inoculum, tempo di incubazione …). Tuttavia, i dati di sensibilità e specificità disponibili suggeriscono che uno qualsiasi dei supporti sopra menzionati può essere utilizzato come routine per il rilevamento di MRSA con elevata affidabilità.
Un altro gruppo di batteri che ha acquisito importanza nel Nosocomiale in infezione sono Enterococchi resistenti alla vancomicina (VRE), fino al punto che il CDC raccomanda lo studio dei vettori in centri di prevalenza elevati. Lo studio dei vettori VRE è stato effettuato utilizzando Sculin Bilis Agar con Azide e Vancomycin (Beav). Il suo utilizzo ha comportato un ritardo diagnostico di 48 ore, oltre alla necessità di eseguire test complementari per una corretta identificazione. Per accelerare questo processo, sono stati sviluppati vari MCS. Uno studio recente confronta cinque mc per VRE utilizzando 400 campioni di feci. I mezzi usati erano Intray Colorx VRE (Biomeddiagnostics, Città Bianca o), Chromid Vre (Biomérieux, Marcyl’étoile, Francia), Vreselect (Bio-rad, Marnes-La-Coquette, Francia), Hardychrom Vre (Hardy Diagnostics, Santa Maria, CA) e Spectra Vre (Remel, Lenexa, KS), utilizzando Beav Agar e Beav Broth come metodo di riferimento. La lettura è stata eseguita a 24 ore. I risultati dei cinque media mostrano una sensibilità compresa tra 89,9 e 94,9%, più alto in tutti i casi di quello mostrato da Agar Beav (84,9%). I migliori dati di sensibilità e specificità continuano a fornirli con il Beav, ma a scapito di un ritardo diagnostico fino a 48 ore rispetto a MC. L’MC più sensibile in questo studio è stato Chromid VR (94,9%), sebbene senza differenze statisticamente significative con il resto.
La specificità e la capacità di differenziare le specie di enterococcus erano simili per i cinque mc. L’intray Colorx VR ha avuto la specificità più bassa con il 98,8% mentre quelle degli altri quattro piatti erano il 99,7%. Le caratteristiche di sensibilità e specificità di tali mezzi lo raccomandano per il rilevamento di vettori VRE quando questo è pertinente. Sono, almeno, perfettamente paragonabili all’agar Beav, diminuendo il tempo di rilevamento a 24-48 h.
Rilevamento di microrganismi gramnegativi
Nel 1979, Kilian e Blow descrivono un nuovo mezzo di coltura che utilizza β-glucoronidasi come a Substrato per il rilevamento diretto di E. coli in urocoltures. Gli studi di redditività hanno indicato che questo metodo ha coinvolto un risparmio economico del 46% e una riduzione del tempo di identificazione del 64% rispetto ai metodi convenzionali39. Da allora, numerosi MCS sono stati sviluppati con diversi substrati per l’identificazione degli uropatologi principali. Uno dei media ha recentemente commercializzato è il Cromid CPS Elite (Biomérieux, Durham, NC) che consente l’identificazione diretta di E. Coli e la presunzione identificativa di Enterococcus spp., Di alcuni enterobacteriaceae e dei batteri del Gruppo Profestore, sebbene in questi casi La conferma dell’identificazione è necessaria da test biochimici o EM Maldi-TOF.
L’efficacia di questo mezzo per il rilevamento di uroopatoogeni43 è stata valutata, valutando il tempo richiesto per la diagnosi e i materiali di consumo utilizzati in confronto a L’uso di agar sangue e agar di macconkey. La concordanza di questo mezzo con la metodologia convenzionale è stata dell’88% per l’urina clinicamente significativa, del 74% per l’urina con una crescita non significativa, il 69% per contaminato e il 95% per le placche senza crescita batterica. Le principali discrepanze erano dovute alla crescita dell’agar del sangue, ma non nella MC dei batteri di GrampoSitivas, in generale con una piccola rilevanza clinica nelle urocolture, come lo Staphylococcus spp. e lactobacillus. Per quanto riguarda il tempo trascorso tra la semina del campione e l’identificazione delle colonie, la differenza non era statisticamente significativa se i due mezzi sono confrontati a livello globale (27.2 h della media per il metodo convenzionale rispetto a 26.6h per il MC), ma era in Quegli orinatoi in cui E. Coli è cresciuto in coltura pura (27.1 h nel metodo convenzionale rispetto a 24,4 h in Chromid, P
0.0001). Gli autori sostengono che questo tempo di miglioramento è dovuto alla facilità e all’affidabilità dell’identificazione del colore, che evita la necessità di metodi di identificazione complementari.
Media Chromid ™ CPS (CPS4) (Biomérieux, St. Laurent, QC) e Uriselect ™ 4 (URS4) (BIO-RAD, Montreal, QC), sono stati valutati44, oltre 903 campioni di urina, rispetto anche al metodo Agar Blood convenzionale e Agar MacConkey. Concordanza con il metodo convenzionale è stato rispettivamente 89,3 e 89,5% per US4 e CPS4. Se solo quei campioni in cui la crescita fosse considerata clinicamente significativa, questa concordanza è aumentata al 93% nel caso dell’URS4 e del 93,1% per il mezzo CPS4.
In tutti i casi, gli autori concordano sul più grande vantaggio di L’uso di MC è la semplificazione del processo di identificazione, riducendo la necessità di test complementari, che consente una diagnosi eziologica più rapidamente.
Data l’utilità dimostrata da questo strumento diagnostico, diversi gruppi stanno progettando un nuovo mc , alcuni non ancora commercializzati, che potrebbero supporre un’alternativa interessante in futuro. Un nuovo Media45 è stato progettato per il rilevamento dei batterini, che include nella sua formulazione di 3,4-cicloexanoscuetine-β-d-glicoside, che è l’obiettivo dell’azione della β-glucosidasi dei batteridi, e che dà origine all’aspetto di Colonie del nero. Questo mezzo è stato confrontato con Agar Sculinear Bilis in 100 campioni fecale. L’MC ha permesso l’identificazione di B. Fragilis in 34 campioni, mentre in Agar Bilis Esculina, questo microrganismo è stato rilevato in 19 campioni. Inoltre, la MC era molto più selettiva, poiché in alcun caso c’era una crescita di specie di non-genere, mentre in agar Bilis Esculina è stata osservata crescita di microrganismi appartenenti ad altri generi (enterobatteri, Clostridium, Candida …) di 34 occasioni. Gli autori studiano anche la capacità di questi MCS, integrati con Meropenem e con metronidazolo, per rilevare la presenza di batteryids resistenti a uno o ad un altro antimicrobico, dimostrando che in entrambi i casi sono in grado di rilevare i ceppi resistenti con una sensibilità del 100% e A Specificità di, almeno l’85%.
Un altro innovativo mezzo sviluppato è il Chromagar EnterocoloCy Yersinia che presenta come un vantaggio rispetto all’ambiente Cessulodine-Irgasan-Novobiocin la possibilità di crescita dei ceppi di Y. Enterocolitica Serovar O3 e Y. Psedotubercolosi, la cui crescita può essere vista inibito nel mezzo CIN46.
Rilevamento del lievito
Sebbene il primo MC sviluppato per funghi, all’inizio degli anni 90 avevano poco successo nella loro applicazione clinica dalla loro limitata capacità di differenziazione, MC per la micologia sviluppata nel Gli ultimi 15 anni hanno avuto un successo molto più grande, poiché consentono una rapida identificazione presuntiva di diverse specie di lievito, uno studio migliore di colture miste e l’identificazione precoce delle specie associate alla resistenza antifunga. I mezzi sviluppati si basano, come nei casi precedenti, in presenza di substrati per uno o più enzimi (β-n-acetil hexosaminidasi e in alcuni casi, β-glucosidasi o fosfatasi), che consentono almeno l’identificazione rapida, da Candida albicans, e in numerosi casi l’identificazione definitiva o presunta di Candida Gabrada, Candida Lusitaniae, Candida Kefyr, C. Parapsilosis, Candida Tropicalis e Candida Krusei. Gli studi disponibili e l’esperienza già prolungata con questo tipo di media dimostrano una specificità di specificità elevata e una buona sensibilità. Tuttavia, data la variabilità del colore e della morfologia che possono apparire in isolati della stessa specie, l’identificazione dovrebbe sempre essere considerata presunta. Un recente studio su oltre 5.600 campioni clinici positivi per il lievito, mostra che oltre l’8% sono stati coltivati misti, il cui rilevamento è molto più probabile se MC viene utilizzato, e propone un algoritmo che combina MC ed EM Maldi-TOF come procedura più appropriata di identificazione47.
Conflitto di interessi
Gli autori dichiarano di non avere alcun conflitto di interessi.