Masas Espectrometría Maldi-Tof. Orixes
A introdución da espectrometría de masas (MS) O tempo de desorción / ionización de ionización láser asistido por matriz (MALDI-TOF) foi, con toda probabilidade, o cambio tecnolóxico do maior borrador ocorreu na clínica de microbioloxía no última década. En poucos anos pasou de ser unha novidade prometedora, para ser unha tecnoloxía totalmente integrada na actividade clínica diaria e dispoñible no noso país, nos servizos de microbioloxía de numerosos centros hospitalarios1. Para iso foi necesario que os espectrómetros de masas sufren unha evolución tecnolóxica significativa. A este respecto, houbo dous progresos que eran cruciales. En primeiro lugar, o deseño do tempo do sistema de voo (TOF) por w.e. Stephens, en 1946, que permitiu a separación de diferentes masas. Ao acelerar os iones nun campo eléctrico e adquirir toda a mesma enerxía cinética, a velocidade que adquiren e, polo tanto, o tempo que se usa para percorrer o tubo de baleiro, depende da masa da molécula ionizada, que se pode inferir do tempo que pasaba Esta ruta.
O segundo fito foi o desenvolvemento de métodos que permitiron as proteínas intactas de ionización que, debido ao seu tamaño, ata entón non fora susceptible á ionización, xa que a alta enerxía necesaria para iso acabou alterando ou destruír a súa propia proteína. En 1987, Köichi Tanaka presenta un novo método de análise (Desorción Láser Soft) que permitiu transferir ás moléculas a enerxía necesaria para ionizas sen romper os fráxiles títulos químicos. Este descubrimento gañoulle o Premio Nobel de Química en 2002, xunto con John B. Fenn, por “o desenvolvemento de métodos de identificación e análise estrutural das macromoléculas biolóxicas”. Non houbo máis espectrómetros de masas baseados neste tipo de análise, aínda que se necesitaban algúns cambios no método para que os em Maldi-Tof acaban por aparecer, no que a desorción dos iones é favorecida por unha matriz que absorbe a enerxía do láser e transferir parcialmente ás moléculas obxecto de estudo. Aínda que estas modificacións melloraron a técnica, facilitándose máis fácil e máis fácil, os responsables destas melloras, Michael Karas e Franz Hillenkamp, non foron incluídos no Premio Nobel, que xerou unha importante controversia. Con base nas contribucións destes autores comezou a ser posible, o estudo das macromoléculas e biopolímeros por MS, que abriu novos campos de solicitude para esta tecnoloxía.
Nos seus case 30 anos de vida, o Maldi EM – TOF foi usado para a análise cuantitativa e cualitativa das proteínas de varias orixes. Inicialmente, aplicouse a proteínas previamente illadas ou pequenos conxuntos de proteínas, pero grazas aos avances técnicos tanto na instrumentación como nas ferramentas para a análise informática dos datos, agora pódese abordar o estudo de grandes grupos de proteínas.
A posibilidade de comezar a estudar proteínas complexas e non só petalpas, ampliando moito os campos potenciais de aplicación de MS. Unha delas foi a identificación de microorganismos que, como o proceso foi simplificado eo software necesarios foron mellorados para explotar os datos en bruto proporcionados polo espectrómetro, axiña deriva na súa aplicación clínica.
MASILLA SPTTROMETRIC Malzi-TOF en microbioloxía clínica
Ata a introdución de Em Maldi-tof, a identificación bacteriana, mesmo con avances significativos como a creación de galerías de identificación miniaturizadas e a automatización da súa inoculación e lectura, continuou a alimentarse polos métodos desenvolvidos pola bacterioloxía clásica A práctica dos sistemas de identificación continuou a baseuse na fermentación de azucres ea súa detección a través do cambio de pH xerado, a metabolización doutros substratos e a produción de diferentes metabolitos e actividades enzimáticas detectables por métodos químicos. Todos estes métodos adulados varias limitacións:
- •
O crecemento bacteriano requirido, que supuxo, na maioría dos casos, un período de incubación de polo menos 16-18 h da súa inoculación ata a lectura.
- •
mostrou problemas de identificación en todos aqueles microorganismos con dificultades para crecer nos medios de comunicación líquidos utilizados para a inoculación destes paneis, así como en microorganismos con baixa actividade bioquímica e enzimática.
- •
Foi necesario considerar a marxe de erro derivada do feito de que, os individuos da mesma especie poden ter diferentes comportamentos contra varios substratos.
Estas limitacións, aínda que eran coñecidas e asumidas, convertéronse en patentes máis claramente cando, dadas as discrepancias observadas nalgúns estudos entre a identificación MSZ-tof e convencional, A secuenciación dos RNR 16S mostrou que, na gran maioría dos casos, a identificación correcta correspondía á proporcionada polos EM MALDI-TOF2.
A este respecto, os em maldi-tof teñen vantaxes obvias:
- •
Independentemente da posibilidade de identificar microorganismos directamente desde algunhas mostras, que se tratan noutra sección, incluso un crecemento precoz ou precoz de placa permiten unha identificación fiable nun curto período de tempo , así aforrar, polo menos, aqueles 16-18 h de crecemento en sistemas de identificación bioquímica.
- •
A análise do perfil de proteína do microorganismo no espectro do 2- 20 kD, que é onde se colocan a maioría das proteínas costelas Osomic, ofrece para a gran maioría das especies bacterianas un perfil específico, o que permite a diferenciación do resto cunha fiabilidade similar á ofrecida pola secuenciación dos RNR 16s.
A isto debe ser engadido que, a introdución desta tecnoloxía, ampliou moito a gama de xéneros e especies que somos capaces de identificar con fiabilidade, cun método capaz de ser usado en rutina. Isto incluso levou a unha reavaliación do papel de patóxenos de microorganismos que, debido á dificultade da súa identificación polos métodos clásicos, probablemente foron infraccións infradiagned3.4.
xa en 1975, Anhalt e Fenselau propón o uso de MS para a identificación de microorganismos5. Vinte anos máis tarde publícase o primeiro estudo que demostra a eficacia da Maldi-TOF EMS para a identificación de microorganismos de células completas6.
En 2009 publicouse que probablemente era un artigo clave para coñecer en xeral , especialistas implicados no diagnóstico de enfermidades infecciosas, as posibilidades de EM MALDI-TOF7. Estuda máis de 1600 illados que inclúen bacterias gramparativas e gramnegativas, aeróbicas e anaeróbicas, obtendo o 95,4% das identificacións correctas. Este artigo está dirixido a este artigo que logo foi transcendido para a utilidade práctica desta metodoloxía: a dispoñibilidade de bases de datos de microorganismos suficientemente amplos, tanto desde o punto de vista cualitativo (número de xéneros e especies incluídos) e cuantitativo (os autores demostran Que a probabilidade de identificación correcta é maior para aqueles microorganismos que existen polo menos dez perfís diferentes introducidos na base de datos).
A partir deste momento, a rápida expansión de uso ocorre desta tecnoloxía na microbioloxía clínica. A primeira publicación en España ten lugar en 2010, mostrando unha correlación coa metodoloxía convencional, a nivel das especies, 100% en gramsitive e 87,7% en gramnegative8. Desde entón, o número de publicacións relacionadas con diferentes aspectos do uso clínico de Em Maldi-tof, pero sobre todo á identificación de microorganismos, foi exponencial7-12.
Identificación de bacterias gramnegativas usando Maldi-tof
Os estudos demostran que a eficiencia na identificación de Enterobacterias e outros gram-negativos é excelente, incluídos os gram-negativos máis comúns en clínica e outros menos frecuentes ou máis complicados de identificar coa metodoloxía clásica (diferentes especies de Yersinia, Aeromonas, Plasiomonas) ,. Brucella, Francisella, Achromobacter, Stenotrophomonas, Burkholderia …). Como noutros casos, os problemas de identificación foron case sempre vinculados a insuficiencias de bases de datos en vez de limitacións do método, como ocorreu nalgúns estudos con xéneros como Ralstonia, Elizabethkingia ou Sphyngobacterium13. Inicialmente, levantáronse algunhas limitacións que puideron ter unha maior transcendencia clínica, como a incapacidade de diferenciar entre Escherichia coli e Shigella14 ou a dificultade de diferenciar Serovars de Salmonella enterica. Os estudos máis recentes suxiren que é posible gardar estas limitacións. Usando programas de ordenador como Flexanalysis e Clinprotools (BRUKER DALTONICS GmbH, Alemaña), identificáronse picos específicos que permiten a diferenza de E. coli e Shigella no 90% dos casos15. Outros autores demostraron que esta diferenciación é posible mesmo sen usar ningún software adicional, simplemente aumentando o número e variedade de perfís de E. coli e Shigella presentes na base de datos de referencia16. Con este método, os autores identifican correctamente 60/64 illados de E. coli e 110/116 illados de Shigella.
a todos aqueles que usaron os EM MALDI-TOF para a identificación, consistía en que, coa metodoloxía habitual, a identificación de Salmonella a un nivel de xénero é fiable, pero máis aló do nivel de xénero é moito Menos. Non obstante, xa hai desde 2004 publicacións que suxiren a existencia de picos específicos que poderían permitir a identificación de serovars con maior fiabilidade17. Un estudo recente suxire a existencia de picos que permiten identificar de forma fiable TYHI18 SEROVAR. Dado que os elementos diferenciais parecen ser existentes, unha extensión da base de datos de referencia cun maior número de altofalantes, polo menos, os serovarios máis frecuentes, probablemente melloraría a resolución neste caso particular.
noutros casos, Como é a diferenciación entre as especies do complexo de Cloacaee Enterobacter, a capacidade dos EM Maldi-Tof é de aproximadamente o 80%, que sen ser óptima, representa unha mellora sensible respecto da metodoloxía convencional19.
no por outra banda, como xa foi mencionado, a identificación correcta de microorganismos anteriormente identificado incorrectamente, como o xénero Raoultella, frecuentemente identificada por métodos clásicos como a Klebsiella ou como un Enterobacter, é permitir unha vista máis real do seu papel como axentes patóxenos, anteriormente undervalued3.
Identificación de bacterias grampositivas usando Maldi-tof
A identificación de microorganismos gram-positivos adoita mostrar figuras algo máis baixas, debido Nalgúns casos á dificultade de mentir a parede, e noutros á similitud entre especies. En xeral, o método directo (aplicación da colonia xunto coa matriz na placa do espectrómetro) adoita ser suficiente para obter unha boa identificación da colonia, pero ás veces a extracción previa con etanol / ácido fórmico pode ser necesaria para conseguir valores de identificación óptimos. Xuntos, ofrece excelentes resultados na identificación de Staphylococcus aureus e outras especies e subespecies de Staphylococos, tanto productores como non productores de coagulasa, así como de EnterocoCCI e outras mortes menos frecuentes como Micrococcus, Gemella, Rothia, etc. Tamén mostrou a súa utilidade na identificación de Bacilli Gram-positivo, incluíndo algunha identificación complexa por métodos clásicos (Listeria, Lactobacillus, Corynebacterium, Nocardia, Actinotignum …) 20.
Ademais, como en O caso dos gramnegativos, o uso sistemático desta tecnoloxía está a cambiar a consideración como patóxenos dalgúns microorganismos, como Staphylococcus Lugdunensis21. Xuntos, os datos dispoñibles fan que sexa aconsellable, no caso de que Staphylococos use, como en calquera outro caso, as bases de datos que ofrecen un maior número de especies e un número razoablemente elevado de perfís por especies e use un método de extracción no lugar de Aplicación directa, ademais de aplicar o microorganismo na placa en duplicado. A redución da puntuación necesaria para validar unha identificación de nivel de especie aos valores > 1.7, en vez de > 2.0 como é común , como algúns autores22 propoñen, poden aumentar o número de identificacións a nivel das especies, pero a cambio dunha diminución da especificidade cuxa relevancia pode ser discutible.
Probablemente o grupo de estreptococos é o que presenta máis problemas entre gramsitivos. Mentres que algunhas das principais especies patóxenas (Streptococcus pyogenes, Streptococcus agalactiae) identificáronse con alta fiabilidade, Streptococcus Pneumoniae (S. Pneumoniae) sempre foi unha fonte de problemas, debido á súa confusión con outras especies do grupo Mitis. Os principais equipos en uso no noso país identifican S. pneumoniae con fiabilidade. O erro máis común é a identificación errónea doutros estreptococos do Grupo Mitis como S. Pneumoniae, especialmente coa base de datos de Bruker23. Algúns picos foron descritos como S. Pneumoniae, polo que algúns autores recomendan o seu uso para corroborar estas identificacións23. Non obstante, a última biblioteca de referencia de Bruker Dalonics (MBT 6903 MSP Biblioteca) segue a recomendar o uso doutras probas (Optoquina, Solubilidade en bilis) para diferencialos.
mesmo dentro do mesmo grupo, a calidade de A identificación das diferentes especies pode variar considerablemente, como ocorre dentro do grupo Streptococcus anginosus.
En calquera caso, como con Staphylococos, o uso dunha base de datos tan ampla como posible e a identificación en duplicado e con protocolos que inclúen a extracción recoméndase optimizar os resultados (fig. 1).
Tramitación grampositivo, gram-negativo, anaerobi e léveda usando maldi-tof.
Identificación de bacterias Anaerobias a través de Maldi-tof
A identificación de bacterias anaeróbicas, tanto grambos como gramnegativas, ten un alto grao de fiabilidade, identificando correctamente as especies máis comúns (bacterias, precipotella, porfiromonas, fusobacterium, clowtridium, actinomyces …). A aplicación dos EM Maldi-Tof neste campo tamén demostrou que a identificación bioquímica era moito menos fiable do que se pensaba que3. A combinación de simplicidade, rapidez e fiabilidade, a continuación, facer os em Maldi-tof no método de elección para a identificación de rutina de bacterias anaeróbicas na clínica. A extracción ou non a mostra non parece modificar significativamente os resultados, nin fai o medio da cultura fonte. Un factor decisivo, como noutros casos, é a amplitude das bases de datos. Xa un dos primeiros estudos realizados en España3 mostrou que a maioría dos erros na identificación de bacterias anaeróbicas foron debido á ausencia de perfís de referencia das especies correspondentes na base de datos. Os estudos máis antigos ofrecen unha eficacia na identificación ao redor do 75-80%, mentres que os estudos máis recentes, con bases de datos máis completas ou bases de datos específicas ofrecen cifras superiores ao 90%. A capacidade de identificar os ribotipos difíciles de Clostridium, suxerido por algúns autores, non se demostrou definitivamente.
Identificación de fungos de léveda e filamentos a través de Maldi-tof
A identificación de léveda foi mostrada desde o inicio excelentes resultados, diferenciando incluso Especies complicadas de discriminación por métodos convencionais, como o complexo de parsilose Candida. Non obstante, os resultados en relación cos fungos filamentosos foron moito máis erráticos. Algúns estudos iniciais mostraron bos resultados estudando selectivamente esporas, pero este método non é práctico nun laboratorio clínico, polo que a maioría dos estudos foron dirixidos ao estudo conxunto de esporas e hifas. No caso de fungos, a extracción convencional con ácido fórmico e acetonitrilo mellora significativamente os resultados. Outros métodos que foron probados, como o uso de contas de vidro para fragmentar as paredes, non parecen ofrecer resultados significativamente superiores á extracción convencional.
Un problema planteado por fungos filamentosos é a existencia de diferenzas significativas Nos perfís de proteínas obtidos segundo a antigüidade das culturas, e mesmo entre a subcultura diferente da mesma CEPA24. A solución a isto pasa pola elaboración de bases de datos máis amplas e máis complexas, que incorporan perfís desde un maior número de cepas e cultivos de diferentes antigüidades. Algúns fabricantes recomendan a eliminación tras unha colleita dun día en caldo, aínda que isto ten un impacto sobre un atraso de 24 h na emisión do resultado. Un estudo realizado en 201125 mostra que unha base de datos ben elaborada e suficientemente complexa é fundamental e que pode mellorar a identificación de fungos filamentosos a cifras similares ás obtido con bacterias e leveduras. Desafortunadamente, a elaboración e validación destas bases de datos son complexas e non están dispoñibles para moitos usuarios, e poucos desenvolvidos están dispoñibles. Polo tanto, e tamén para fins de estandarización, probablemente sexa máis apropiado usar as bases de datos dos fabricantes, que deben ser expandidos convenientemente.
Actualmente Bruker DalTonics, cuxa base de datos de fungos filamentosos, na súa versión MBT 6903 MSP Biblioteca , inclúe 25 xéneros e 42 especies, recomenda a cultura en medio líquido durante a noite, seguido de centrifugación, lavado e extracción con etanol, ácido fórmico e acetonitrilo (Fig. 2). MS-Vitek, na súa versión 3.0, inclúe 32 xéneros e 81 especies, e Saramis na súa versión RUO 4.13, inclúe unha base de datos máis ampla, con 45 xéneros e 168 especies. Biomérieux non recomenda a cultura en medio líquido, senón unha extracción convencional con etanol, ácido fórmico e acetonitrilo (Figura 2). A pesar de todo, un estudo recente utilizando a metodoloxía recomendada por Bruker identifícase correctamente, a nivel de especies, só o 72% do insulado26 e un estudo moi recente con Vitek 3.0 identifica correctamente o 66,8% de 318 illados, debido especialmente ás deficiencias da base de datos27. Polo tanto, hoxe, a identificación de fungos filamentosos por em Maldi-tof aínda non pode substituír completamente a metodoloxía convencional de identificación.
Procesamento Micobacterial recomendado para o sistema Biotyper MALDI.
4895790f6a “>
Identificación de Mycobacteria usando Maldi-tof
As limitacións da metodoloxía tradicional para a identificación de Mycobacteria, sobre todo en que se refire ao tempo de resposta, eles Fixeron a práctica a totalidade dos laboratorios decantáronse polos métodos de identificación molecular. O uso de EM Maldi-Tof para a identificación de Mycobacteria é unha alternativa igualmente rápida e máis barata que as técnicas moleculares, pero mostra peculiaridades en varios aspectos. Por unha banda, os métodos directos de análise, ou que implican unha alta concentración ou manipulación dos microorganismos previamente á súa inactivación, non son recomendados por unha cuestión de seguridade biolóxica. O uso dun método de extracción que garante a ruptura das células mellora a calidade dos espectros e aumenta a seguridade do procedemento.
Varios factores poden influír na eficacia da identificación de Mycobacteria por Em Maldi – Tof. Como tamén se mostra con fungos, Mycobacteria pode ter perfís de proteínas moi diferentes en función da antigüidade de cultivos, polo que é importante que o diagnóstico inclúa perfís de proteínas de cultivos de diferentes antigüidades para cada microorganismo. Tamén se falou sobre a posible formación de polímeros que enmascaran o tamaño real das proteínas características utilizadas para a identificación. Todo isto fai a identificación de Mycobacteria un procedemento menos previsible en función dos seus resultados que a doutras bacterias, como demostra a heteroxeneidade dos datos publicados. Nas últimas actualizacións, as bibliotecas experimentaron importantes melloras. Así, por exemplo, a biblioteca de Micobacterial de Biotyper inclúe 149 especies e a versión 3.0 de Vitek MS incorpora 48 novas especies con respecto á versión anterior.
O procedemento inicialmente recomendado por Bruker incluíu unha serie de pasos (axustándose a Unha determinada densidade óptica, varios pasos de centrifugación e resuspensión) que asumiron un risco biolóxico probablemente innecesario, polo que foron substituídos por un único paso de tratamento con calor. O método recomendado actualmente inclúe varios pasos de calefacción e lavado, seguido dun tratamento con bolas de sílice e acetonitrilo. Posteriormente, son tratados con ácido fórmico, aplicado na placa e cubertos coa matriz (Fig. 3). Pola contra, o método defendido pola parte biomérieux dun tratamento inicial con 70% de bolas de etanol e sílice, despois de que a mostra extrae con ácido fórmico e acetonitrilo, aplicada na placa e cuberta coa matriz (fig. 4).
Procesamento Micobacterial recomendado para o sistema Vitek MS.
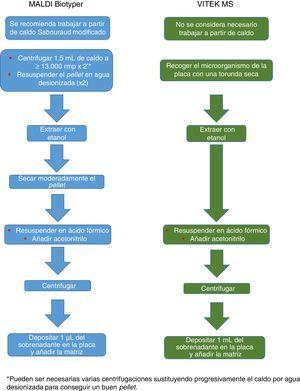
Procesamento de fungos Recomendado para Maldi Biotyper e Vitek MS Systems.
un estudo comparativo entre ambos métodos realizados recentemente NTE28 mostra que aínda que a identificación tende a ser algo mellor a partir da metade de 7h10 que a Lowenstein Jensen, as diferenzas son pequenas en ambos sistemas. Non obstante, demostra que o método de procesamento da mostra é crítico e non é intercambiable entre os sistemas. As mostras obtidas co método de extracción de brúra proporcionan mellores resultados no seu espectrómetro e os procesados co método Vitek faino con salamis e vitek a si mesma, aínda que a diferenza neste caso é menor. Xuntos, Vitek MS parece estar menos afectado polo método de extracción. En canto á eficiencia na identificación, Bruker Biotyper identificou unha puntuación №1,8 81,5% dos illados, mentres que Saramis e Vitek MS 3.0 identificáronse cunha fiabilidade ≥90% en 85,4 e un 89,2% respectivamente. Usando estes mesmos criterios, o 22,6% dos illados que foron identificados por Bruker Biotyper necesitaban ser extraídos en máis dunha ocasión para lograr a identificación, que ocorreu un 21,5% con Saramis e un 14,2% con Vitek MS 3.0.
En relación a outros microorganismos con estrutura parietal próxima á de Mycobacteria, polo tempo considerouse que a súa identificación foi problemática debido á composición do seu muro. Non obstante, demostrouse que, se unha base de datos está dispoñible con referencias suficientes, un método de extracción similar ao usado con Mycobacteria pode ofrecer bos resultados29.
Todos os datos referidos anteriormente suxiren que o espectro de microorganismos susceptibles A identificación por Em Maldi-Tof é extraordinariamente ampla e que, con excepcións, o factor limitante da capacidade do Maldi-Tof emite case sempre máis na integridade das bases de datos de referencia que a capacidade do método para obter perfís confiables de case calquera microorganismo bacteriano capaz de crecer en medios de cultivo. De feito, as empresas de fabricación parecen ser conscientes do problema; Así, a última biblioteca de referencia publicada por Bruker Daltonics (MBT 6903 MSP Biblioteca) incorpora 938 novos perfís, dos cales 17,2% están destinados a cubrir novos xéneros e especies (15 novos xéneros e 96 especies novas especificamente), en particular Anaerobios e fungos, Pero o 82,8% está destinado a aumentar a diversidade de perfís dentro das especies xa contempladas en bibliotecas anteriores. Do mesmo xeito, VITEK MS versión 3.0 inclúe 242 novas especies bacterianas, entre as que hai 48 novas micobacterias e 15 noites e 55 novos fungos, cunha media global de máis de 10 espectros illados e 25 por especies.
Identificación de microorganismos, por maldi-tof, desde a mostra directa
a posibilidade de identificación de microorganismos antes do crecemento das colonias foi, desde o principio, un obxectivo moi atractivo, polo que supón o momento de poder establecer un O tratamento empírico máis orientado, reduce a aparición da resistencia e optimiza o gasto30. Os EM Maldi Tof, aplicados a unha mostra directa, converteuse nunha ferramenta moi útil en infeccións graves con alta taxa de morbilidade como a bacteremia e as fungemias, aínda que o traballo directo sobre as mostras ten inconvenientes que poden limitar a utilidade da técnica. En mostras con baixa densidade microbiana non é posible realizar unha identificación correcta, xa que a cantidade de proteínas bacterianas presentes é insuficiente. Algo similar ocorre coas mostras poliméticas, xa que se pode xerar un espectro de proteína aberrante, como resultado da mestura de varios perfís ou ignorando directamente o microorganismo que está nunha menor proporción. Desenvolvéronse algunhas melloras de software, que ás veces poden detectar e incluso identificar os microorganismos implicados por separado, pero require ser coidadosamente estudado e probablemente refinado a ter unha utilidade real. Na maioría dos métodos que traballan directamente na mostra, e antes da posible presenza de microorganismos intracelulares, é aconsellable empregar métodos que inclúen a lise celular.
Non todas as mostras son válidas para este tipo de estudo. A mostra ideal é a que provén dunha área habitualmente estéril, que pode acomodar altas concentracións de microorganismo e en que non hai limitacións significativas sobre o volume de mostra. As mostras de áreas habitualmente colonizadas (pel, feces, aparellos respiratorios altos …) xerarán perfís aberrantes en toda probabilidade. Un tema técnico para ter en conta cando se traballa directamente na mostra é o volume de mostra dispoñible, que en casos como a orina ou as culturas de sangue non adoita ser un problema, senón que noutros con fluído cerebrospinal pode ser, e que será como Moito máis importante como menor é a concentración bacteriana. Ademais, as mostras con alto contido de proteína da fonte alieníxena ao microorganismo tamén poden xerar problemas no momento da súa interpretación31.
Con todo, a vantaxe que é, especialmente nas hemoculturas, superar a 24 horas da identificación de O microorganismo e orientación do tratamento máis específicamente, fixo o uso directo de MS na mostra, especialmente nestes casos, ten xeneralizado.
A identificación directa das mostras de orina
A orina é unha das mostras que mellor se axustan ao Condicións ideais para traballar con EM Maldi-Tof en mostra directa. É un fluído estéril baixo condicións fisiolóxicas, a carga bacteriana na orina infectada é alta na maioría dos casos e as limitacións sobre o volume de mostra e obtención de novas mostras se é necesario, son mínimas.
Estudos con excelente Os resultados foron publicados, con concordancia na identificación que alcanza o 90-95% 32.33 en comparación cos sistemas automatizados.Describíronse diferentes protocolos de procesamento de mostras, todos eles coinciden coa necesidade dunha selección de pre-selección (citometría de fluxo, gramo) para discriminar entre mostras positivas e negativas e limitar o uso de MS a samples positivos inicialmente34.
Con pequenas variacións, a preparación das mostras adoita consistir en envialas a unha serie de centrifugacións e lavadas con auga desionizada, a continuación, realizando o procedemento convencional do Maldi-Tof. Modificacións foron feitas incorporando a mostra SDS ALIQUOT a 10% 33 para mellorar a liberación de proteínas ou tween-8034 para mellorar os resultados. Os resultados obtidos adoitan ser mellores con gramnegativos que con grampositivos e fungos, mellorando a fiabilidade cando os contas son altos, por riba de 105fc / ml.
Un inconveniente deste método foi a necesidade de realizar a Estudo de sensibilidade a través da metodoloxía convencional. Un estudo recente suxire que, unha vez que a identificación de Em Maldi-tof, o mesmo sedimento pode usarse para realizar un disco de disco anticorado con excelentes resultados, o que permite reducir o 24 h o estudo completo35. O importante cambio organizativo que supón a introdución desta metodoloxía e as infeccións nas que, moitas veces, as poucas horas son diagnosticadas non é fundamental para a xestión do paciente, fixeron que o diagnóstico de infección urinaria a través de Em Maldi-Tof non tivo a Penetración que tivo, por exemplo, no caso das culturas sanguíneas. É necesario alcanzar unha maior sensibilidade e unha maior normalización dos métodos a considerar a súa introdución na rutina clínica.
Identificación directa a partir de hemoculturas
O establecemento do tratamento empírico correcto é decisivo na evolución da bacteriemia A probabilidade de que o tratamento establecido sexa correcto, será maior, a información máis precisa e concreta que somos capaces de proporcionar rapidamente. Neste aspecto, a posibilidade ofrecida polos EM Maldi-tof de obter unha identificación fiable nun curto período de tempo despois da positivización da cultura sanguínea é un avance evidente.
Numerosos protocolos de procesamento cultural-cultural-sangue teñen Describiuse (centrifugación diferencial, lise celular e extracción de proteínas por métodos químicos, separación de gel …). A maioría deles baséanse na lise e eliminación de compoñentes celulares, xa sexa por centrifugacións e lavados, nos que se usan diferentes compostos como saponina, cloruro de amonio ou SDS ou usando tubos con gel de separación de soro e activador de coagulación. En definitiva, o que se busca con estes métodos é illar e concentrar microorganismos ata alcanzar polo menos 105-107FC / ml, concentración á que a cantidade de proteínas é suficiente para xerar perfís axeitados en Maldi-tof.
No caso das hemoculturas hai un procedemento comercial aprobado (Sepsityper, Bruker Daltoniks GmbH, Alemania), que consta da adición dunha solución de lisis a unha aliqueca de 1ml da cultura sanguínea, despois de que a mostra está suxeita a varios pasos de lavado e centrifugación e, finalmente, nunha extracción convencional con etanol e ácido fórmico36.
Os resultados do procesamento directo das culturas de sangue son xeralmente boas, con porcentaxes de identificación correctas de 80-90%, aínda que con algúns matices. A identificación de gram-negativos adoita ser correcta no 90-95% dos casos, mentres que en gramsitivos é moito máis heteroxéneo, oscilando entre figuras similares ás de Gram-negativas e figuras ao redor do 50%. Dentro destes, representan problemas de fiabilidade sobre todos os estreptococos do Grupo Viridans e non coagulas de Staphylocococos non productores.
Os resultados dos casos de fungos, nos primeiros estudos foron decepcionantes. Unha extracción correcta, indispensable neste caso, conduce a figuras de identificación superiores ao 90%, homologadas coas obtenidas en bacteremias37. Pódese ter en conta o posible impacto de determinados compoñentes dalgúns hemocultivos, como o carbono activo, que pode interferir coa identificación. Do mesmo xeito, un tempo de incubación prolongada da cultura sanguínea pode ter un impacto negativo sobre os espectros obtidos. Non obstante, dado que o procedemento habitual é realizar a MS, como máximo, unhas horas despois da positivización e máis do 80% das culturas sanguíneas positivas significativas están nos primeiros 48 h, o impacto clínico deste aspecto non parece transcendente ..
Ao avaliar a heteroxeneidade das porcentaxes de identificación correctas nas hemoculturas, hai que ter en conta que non hai criterio homoxéneo sobre como avaliar as puntuacións obtidas. Algúns autores consideran a identificación correcta cos valores > 1.7. Outros reducen este requisito de ata 1,5, pero inclúen requisitos como a identificación repítese nas dúas ou tres primeiras posicións da posible lista de identificación ou que entre as dúas primeiras opcións que ofrece a diferenza de Maldi-tof hai unha diferenza de puntuación de polo menos 0,3 puntos.
En xeral, non estamos a favor de reducir o nivel de requisito na identificación correcta nas culturas de sangue, xa que a existencia dunha maior porcentaxe de “sen identificacións” supoñerá sempre un risco menor, desde o punto de vista clínico, que a proliferación de identificacións incorrectas, que poderían condicionar tratamentos inadecuados.
No que se refire ao uso de métodos de procesamento comercial, como sepsityper, os resultados en xeral son similares en relación cos métodos de procesamento manual. Cada laboratorio debe priorizar entre a maior normalización e aforro de tempo de procesamento que propiciou estes métodos ou aforro económico (en torno a 1 / mostra) que implica o uso dun método manual.
en conxunto, em maldi- TOF é un método rápido e fiable para a identificación directa dos microorganismos en hemoculturas. A súa combinación con métodos que permiten a detección de mecanismos de resistencia a certos antimicrobianos permiten ofrecer información clínica valiosa a un tempo significativamente menor (24-48 h menos) segundo o requiren a metodoloxía convencional. Probablemente o punto principal para elucidar, neste momento, é como integrar esta nova actividade en laboratorios de microbioloxía, polo que a información ofrecida está optimizada sen supoñer unha sobrecarga por servizos, normalmente non precisamente sobre o técnico de persoal e con estruturas de tempo moi diverso.
Identificación directa doutras mostras
O número de estudos relacionados coa aplicación directa do Mali-tof em a outras mostras biolóxicas é menor. Como se discutiu noutra sección, a utilidade de Em Maldi-tof nestas mostras está determinada por varios factores: por unha banda, a mostra debe provir dunha área estéril baixo condicións fisiolóxicas e en que a infección, cando apareza, almacena ser xeralmente monomicrobial. Doutra banda, tanto a carga bacteriana presente na mostra son cruciales, como o volume de mostra dispoñible para o estudo. Isto fai que moitas mostras de urina e hemoculturas poidan proporcionar problemas, por baixo volume que normalmente están dispoñibles (CSF, exudativos purulentos), debido á baixa concentración de microorganismos (CSF, fluído peritoneal en pacientes que sufriron diálise peritoneal) ou por presenza de importantes cantidades de non -Proteínas bacterianas que poden alterar os perfís ou interferir coa ionización das proteínas bacterianas (exudativos purulentos).
baixa sensibilidade nestes produtos nos que a carga bacteriana non é moi alta e o volume de mostra dispoñible, Moitas veces limitadas, fan que as técnicas moleculares sexan preferibles para o diagnóstico rápido, mesmo coa limitación que a gama de microorganismos detectables nunha única proba é moito menor que con eles cuzii -tof. Os diagnósticos etiolóxicos de meninxite bacteriana ocasionalmente comunicaron por cdri-tof en CSF, pero un estudo informado a 21ereccmid indicou que, en 183 mostras de CSF, das cales 14 foron positivas para o método convencional, ningún foi positivo por EM cuzid -tof directamente Sample38, que reafirma o feito de que, desde o punto de vista da sensibilidade, a Maldi-Tof em non pode competir coas técnicas moleculares. Noutros tipos de mostra, a experiencia é moi limitada, non hai protocolos ou datos publicados sobre sensibilidade e especificidade e, polo tanto, o uso clínico de EM MALDI-TOF, neste momento, non é pertinente.
Media Chromogenic
Desde a aparición do primeiro medio cromoxénico (MC), hai máis de 30 anos, converteuse nunha ferramenta moi útil para o illamento diferencial dos microorganismos patóxenos. Actualmente hai multitude MC comercializada pola identificación de bacterias e fungos, así como para o estudo dalgúns mecanismos de resistencia antimicrobiana.
A fundación destes medios na inclusión dun substrato cromogénico que, sendo hidrolizado por Un enzimático específico presente no microorganismo, dá lugar a unha colonia de colonación característica, permite a súa diferenciación.Tamén son tamén medios selectivos, que inhiben o crecemento doutros microorganismos en maior ou extensión, favorecendo a detección de colonias de cores. Os substratos principais do MCS comercial son derivados indólicos39 que poden ser hidrolizados por galactosidas ou glucosidases, producindo derivados tóxicos baixos e que non inhiben o crecemento bacteriano.
dedicáronse a dedicarse a O desenvolvemento de métodos para a detección de resistente a meticilina S. aureus (MRSA), tendo en conta a súa importancia na infección nosocomial. Neste contexto, o uso de MC recolleu gran importancia para o rápido illamento e identificación desta bacteria. Hai numerosos MC no mercado para a detección de MRSA. Un estudo recente40 compara tres deles: Chromid MrSA Smart (SMART), Chromid MRSA Fist Generation (Chromid) e Brilliance MRSA (OX2) para a selección de 1.220 mostras de pacientes hospitalizados. A detección destes medios foi corroborada por EM Maldi-Tof, difusión en Agar con discos Cefoxitina e PCR Comercial para Meca e MecC. A sensibilidade ás 24 h era mellor para o medio intelixente con respecto ao medio cromo, pero non houbo diferenzas significativas co ambiente de OX2. Non obstante, os autores indican que aínda é necesario, para obter resultados óptimos, o enriquecemento de caldo por 24 h antes da inoculación en calquera dos medios estudados.
Xu et al.41 Comentarios de 5 dos medios máis usados para Detección MRSA. O que mellor resulta neste estudo é o Brilliance MRSA 2 (OXOID LTD, TERMOFISE, EUA), cunha sensibilidade do 65,7% e unha especificidade do 99,8%. O rendemento deste medio mellora se a mostra está inoculada nun caldo de enriquecemento antes de plantalo no MC, cunha sensibilidade do 100% e unha especificidade do 99,1%. Os medios de comunicación Cromagar MRSA mostra unha sensibilidade do 95% despois das 24 horas da incubación, que alcanza o 100% se se incuba 48 h. A especificidade en ambos casos é do 100%. O mediano Agar Agar Cromo MRSA inclúe a Cefoxitina na súa composición, que simplifica a interpretación dos resultados. Este medio foi estudado usando illados MRSA obtidos a partir de hemoculturas, cunha sensibilidade e especificidade de 97,6 e 99% respectivamente. A selección MRSA foi evaluada usando 652 illados a partir de hemoculturas despois de 18-24 h de incubación a 35 ° C, cunha sensibilidade do 99% e unha especificidade do 98%, o que aumentou un 99% cando se combina coa proba de coagula. O cromo medio MRSA, que tamén incorpora a cefoxitina na súa composición, foi estudada en illados de lesións e cultivos de sangue. Nas hemoculturas mostra unha sensibilidade do 97,8% ás 24 h, o que aumenta o 100% ás 48 h e unha especificidade do 99,7% que se mantén (99,6%) ás 48 h. En feridas, a sensibilidade e a especificidade foron de 88,9 e 100% a 24 h e 100% en ambos casos ás 48 h.
O establecemento de comparacións entre este tipo de medios de cultura é complexo, xa que hai moitos factores, Ademais do medio de cultivo, que pode influír nos resultados (tipo de mostra, concentración de inóculo, tempo de incubación …). Non obstante, os datos de sensibilidade e especificidade dispoñibles suxiren que calquera dos medios mencionados anteriormente pode ser usado como unha rutina para a detección de MRSA con alta fiabilidade.
Outro grupo de bacterias que adquiren importancia na infección son nosocomiales Enterococci (VRE) resistente á vancomicina (VRE), ata o punto de que o CDC recomenda o estudo das operadoras en centros de prevalencia elevados. O estudo de VRE Carriers realizouse usando Sculin Bilis Agar con Azide e Vancomycin (BEAV). O seu uso implicaba un atraso de diagnóstico de 48 h, ademais da necesidade de realizar probas complementarias para a identificación adecuada. Para acelerar este proceso, desenvolvéronse varios MCS. Un estudo recente compara cinco MC para VRE usando 400 mostras de feces. Os medios utilizados foron intray colorx vre (BiomedDiagnostics, White City, OR), chromID VRE (bioMérieux Marcyl’étoile, Francia), Vreselect (Bio-Rad, Marnes-la-Coquette, Francia), Hardychrom VRE (Hardy Diagnostics, Santa Maria, CA) e Spectra VRE (Remel, Lenexa, KS), usando Beav Agar e Beaav Broth como método de referencia. A lectura realizouse ás 24 h. Os resultados dos cinco medios mostran unha sensibilidade entre 89,9 e 94,9%, maior en todos os casos que o mostrado por Agar BEAV (84,9%). Os mellores datos de sensibilidade e especificidade continúan proporcionándolles o BEAV, pero a costa dun atraso de diagnóstico de ata 48 h con respecto a MC. O MC máis sensible neste estudo foi Chromid VR (94,9%), aínda que sen diferenzas estadísticamente significativas co resto.
A especificidade e capacidade de diferenciar as especies de enterococcus foron similares para os cinco MC. O Intray Colorx VR tiña a especificidade máis baixa cun 98,8% mentres que os doutras catro placas foron do 99,7%. As características de sensibilidade e especificidade destes medios fan que se recomenda a detección de transportistas VRE cando isto sexa pertinente. Son, polo menos, perfectamente comparables co Agar BEAV, diminuíndo o tempo de detección a 24-48 h.
Detección de microorganismos gramnegativos
En 1979, Kilian e golpe describen un novo medio de cultivo que usa β- glucoronidasa como a Substrate para a detección directa de E. coli en Urocultures. Os estudos de rendibilidade indicaron que este método implicou un aforro económico do 46% e unha redución do 64% de tempo de identificación con respecto aos métodos convencionais39. Desde entón, numerosos MC foron desenvolvidos con diferentes substratos para a identificación dos principais uropatóxenos. Un dos medios de comunicación recentemente comercializado é Chromid CPS Elite (biomérieux, Durham, NC) que permite a identificación directa de E. coli, ea identificación presuntiva de Enterococcus spp., Dalgunhas enterobacteriaceae e as bacterias do grupo Proveae, aínda que nestes casos A confirmación da identificación é necesaria por probas bioquímicas ou em Maldi-tof.
A eficacia deste medio para a detección de ioopathogens43 foi avaliada, avaliando o tempo necesario para o diagnóstico e consumibles utilizados en comparación con O uso de Agar Slood e Macconkey Agar. A concordancia deste medio coa metodoloxía convencional foi do 88% para a orina clínicamente significativa, do 74% para a orina con crecemento non significativo, o 69% para a contaminación eo 95% para as placas sen crecemento bacteriano. As discrepancias principais foron debido ao crecemento do agar sangue, pero non no MC das bacterias grampositivas, en xeral con pouca relevancia clínica en uroculturas, como Staphylococcus spp. e lactobacillus. En canto ao tempo transcorrido entre a sementeira de mostra ea identificación das colonias, a diferenza non foi estadísticamente significativa se os dous medios compáranse globalmente (27,2 horas de media para o método convencional en comparación con 26,6 horas para o MC), pero estaba en Aqueles urinarios nos que E. coli creceu en cultura pura (27,1 h no método convencional en comparación con 24,4 h en Chromid, P
0,0001). Os autores argumentan que este tempo mellorado débese á facilidade e fiabilidade da identificación de cores, que evita a necesidade de métodos de identificación complementarios.
Media Chromid ™ CPS (CPS4) (Biomérieux, St. Laurent, QC) e Uriselect ™ 4 (URS4) (Bio-Rad, Montreal, QC), foron avaliados44, máis de 903 mostras de orina, en comparación tamén co método de agar de Agar e Macconkey convencional. A concordancia co método convencional foi de 89,3 e 89,5% para Urs4 e CPS4 respectivamente. Se só esas mostras en que o crecemento considerouse clínicamente significativo, esta concordancia aumentou ao 93% no caso de URS4 e 93,1% para o medio CPS4.
En todos os casos, os autores coinciden en que a maior vantaxe de O uso de MC é a fusilación do proceso de identificación, reducindo a necesidade de probas complementarias que permite un diagnóstico etiolóxico máis rápido.
Dada a utilidade demostrada a partir desta ferramenta de diagnóstico, varios grupos están deseñando novo MC , algúns aínda non comercializados, que poden supoñer unha alternativa interesante no futuro. Un novo MEDIA45 foi deseñado para a detección de bacterias, que inclúe na súa formulación de 3,4 ciclohexanoscuetine-β-D-glicósida, que é o obxectivo de acción da β-glucosía de bacterias e que dá orixe á aparición de Colonias de negro. Este medio foi comparado co Agar Sculinear Bilis en 100 mostras fecais. O MC permitiu a identificación de B. fragilis en 34 mostras, mentres que en Agar Bilis Esculina, este microorganismo foi detectado en 19 mostras. Ademais, o MC era moito máis selectivo, xa que en ningún caso foi un crecemento de especies de xénero, mentres que no Agar Bilis esculina observouse crecemento de microorganismos pertencentes a outros xéneros (Enterobacteria, Clostridium, Candida …) por 34 ocasións. Os autores tamén estudan a capacidade destes MCS, complementados con meropenem e con metronidazol, para detectar a presenza de bacterias resistentes a unha ou outra antimicrobiana, demostrando que en ambos casos son capaces de detectar cepas resistentes cunha sensibilidade do 100% e a especificidade de, polo menos o 85%.
Outro medio innovador desenvolvido é o Chromagar enterocolicy Yersinia que presenta como unha vantaxe sobre o ambiente Cessulodine-Irgasan-Novobiocin a posibilidade de crecemento das cepas de Y. Enterocolitica Serovar O3 e Y. Psedotuberculosis, cuxo crecemento pode verse inhibido no medio cin46.
Detección de levadura
Aínda que o primeiro MC desenvolveu para fungos, a principios dos 90, tiñan pouco éxito na súa aplicación clínica pola súa capacidade de diferenciación limitada, MC para a micoloxía desenvolvida no Os últimos 15 anos tiveron un éxito moito maior, xa que permiten unha rápida identificación presuntiva de diferentes especies de levadura, un mellor estudo de cultivos mixtos e a identificación precoz das especies asociadas á resistencia antifúngica. Os medios desenvolvidos están baseados, como nos casos anteriores, en presenza de substratos por unha ou varias enzimas (β-n-acetil hexosaminidasa e, nalgúns casos, β-glucosía ou fosfatase), que permiten identificación rápida, polo menos, polo menos, Por Candida Albicans, e en numerosos casos a identificación definitiva ou presunta de Candida Gabrada, Candida Lusitaniae, Candida Kefyr, C. Parapsilose, Candida Tropicalis e Candida Krusei. Os estudos dispoñibles e a experiencia xa prolongada con este tipo de medios demostran unha especificidade de alta especificidade e boa sensibilidade. Non obstante, dada a variabilidade da cor e a morfoloxía que pode aparecer en illados da mesma especie, a identificación sempre debe considerarse presuntivo. Un estudo recente sobre máis de 5.600 mostras clínicas positivas para a levadura, mostra que máis do 8% foron cultivos mixtos, cuxa detección é moito máis probable se o MC sexa usado e propón un algoritmo que combine MC e EM Maldi-tof como procedemento máis apropiado de identificación47.
Conflito de interese
Os autores declaran non ter ningún conflito de intereses.