Espectrometria de masses MALDI-TOF. Orígens
La introducció de l’espectrometria de masses (EM) matrix-assisted laser desorption / Ionization time of flight (MALDI-TOF) ha estat, amb tota probabilitat, el canvi tecnològic de major calat esdevingut en la Microbiologia clínica a l’última dècada. En pocs anys ha passat de ser una prometedora novetat, a ser una tecnologia totalment integrada en l’activitat clínica diària i disponible, a casa nostra, als Serveis de Microbiologia de nombrosos centres hospitalarios1. Per a això ha estat necessari que els espectròmetres de masses patissin una important evolució tecnològica. Referent a això, va haver-hi dos avanços que van resultar crucials. En primer lloc, el disseny de sistema Time of Flight (TOF) per W.E. Stephens, el 1946, que va permetre la separació de masses diferents. A l’accelerar els ions en un camp elèctric i adquirir tots la mateixa energia cinètica, la velocitat que adquireixin, i per tant el temps emprat en recórrer el tub de buit, depèn de la massa de la molècula ionitzada, que podrà ser inferida a partir de l’ temps emprat en aquest recorregut.
la segona fita va ser el desenvolupament de mètodes que permetien ionitzar proteïnes intactes que, per la seva grandària, fins llavors no havien estat susceptibles d’ionització, ja que l’alta energia requerida per a la mateixa acabava alterant o destruint la pròpia proteïna. El 1987, Koichi Tanaka presenta un nou mètode d’anàlisi (Soft Laser Desorption) que permetia transferir a les molècules l’energia necessària per ionizarlas sense trencar els fràgils enllaços químics. Aquest descobriment li va valer el Premi Nobel de Química el 2002, juntament amb John B. Fenn, per “el desenvolupament de mètodes d’identificació i d’anàlisi estructural de macromolècules biològiques”. No van trigar a desenvolupar-espectròmetres de masses basats en aquest tipus d’anàlisi, encara que van ser necessaris alguns canvis en el mètode perquè finalment aparegués l’EM MALDI-TOF, en la qual la desorció dels ions és afavorida per una matriu que absorbeix l’energia de l’ làser i la transfereix parcialment a les molècules objecte d’estudi. Tot i que aquestes modificacions van millorar la tècnica, fent-la més senzilla i sensible, els responsables d’aquestes millores, Michael Karas i Franz Hillenkamp, no van ser inclosos en el premi Nobel, la qual cosa va generar una important controvèrsia. A partir de les aportacions d’aquests autors va començar a ser possible l’estudi de macromolècules i biopolímers mitjançant EM, el que va obrir nous camps d’aplicació per a aquesta tecnologia.
En els seus gairebé 30 anys de vida, l’EM MALDI -TOF s’ha utilitzat per a l’anàlisi quantitativa i qualitativa de proteïnes d’orígens diversos. Inicialment es va aplicar a proteïnes aïllades prèviament o petits conjunts de proteïnes, però gràcies als avenços tècnics tant en la instrumentació com en les eines per a l’anàlisi informàtic de les dades, actualment es pot abordar l’estudi de grans grups de proteïnes.
la possibilitat de començar a estudiar proteïnes complexes, i no només petits pèptids, va ampliar molt els camps potencials d’aplicació de l’EM. Un d’ells va ser la identificació de microorganismes que, a mesura que es va simplificar el procediment i es va millorar el programari necessari per explotar les dades crues proporcionades per l’espectròmetre, va derivar ràpidament en la seva aplicació clínica.
Espectrometria de masses MALDI-TOF en Microbiologia Clínica
Fins a la introducció de l’EM MALDI-TOF, la identificació bacteriana, fins i tot amb avanços significatius com la creació de galeries d’identificació miniaturitzades i l’automatització de la seva inoculació i lectura, seguia nodrint-se dels mètodes desenvolupats per la bacteriologia clàssica. La pràctica totalitat dels sistemes d’identificació seguien basant-se en la fermentació de sucres i la seva detecció mitjançant el canvi de pH generat, la metabolització d’altres substrats i la producció de diferents metabòlits i activitats enzimàtiques detectables per mètodes químics. Tots aquests mètodes patien de diverses limitacions:
- •
Requerien creixement bacterià, el que suposava, en la major part dels casos, un període d’incubació de al menys 16-18 h des seva inoculació fins a la seva lectura.
- •
Mostraven problemes d’identificació en tots aquells microorganismes amb dificultats per créixer en els mitjans líquids usats per a la inoculació d’aquests panells, així com en microorganismes amb escassa activitat bioquímica i enzimàtica.
- •
Era necessari considerar el marge d’error derivat de el fet que, individus de la mateixa espècie, poguessin tenir comportaments diferents davant de diversos substrats.
Aquestes limitacions, tot i que eren conegudes i assumides, han quedat més clarament patents quan, davant les discrepàncies observades en alguns estudis entre l’EM MALDI-TOF i la identificació convencional, la seqüenciació de l’ARNr 16S va demostrar que, en la gran majoria dels casos, la identificació correcta corresponia a la proporcionada per l’EM MALDI-TOF2.
Referent a això, l’EM MALDI-TOF té avantatges evidents:
- •
Amb independència de la possibilitat d’identificar microorganismes directament a partir d’algunes mostres, que es tracta en un altre apartat, creixements en placa fins i tot molt escassos o precoços permeten obtenir una identificació fiable en un curt període de temps, estalviant així, com a mínim, aquestes 16-18 h de creixement en els sistemes bioquímics d’identificació.
- •
l’anàlisi de l’perfil proteic de l’microorganisme al espectre dels 2-20kD, que és on se situen la major part de les proteïnes rib osómicas, ofereix per a la gran majoria de les espècies bacterianes un perfil específic, que permet diferenciar-les de la resta amb una fiabilitat semblant a la que ofereix la seqüenciació de l’ARNr 16S.
A això cal afegir el fet que, la introducció d’aquesta tecnologia, ha ampliat molt el ventall de gèneres i espècies que som capaços d’identificar amb fiabilitat, amb un mètode susceptible de ser usat en rutina. Això ha portat fins i tot a una reavaluació de el paper com patògens de microorganismes que, per la dificultat de la seva identificació pels mètodes clàssics, estaven molt probablement infradiagnosticados3,4.
Ja el 1975, Anhalt i Fenselau proposen la utilització de l’EM per a la identificació de microorganismos5. Vint anys després es publica el primer estudi que demostra l’eficàcia de l’EM MALDI-TOF per a la identificació de microorganismes a partir de cèl·lules completas6.
El 2009 es va publicar el que probablement va ser un article clau per donar a conèixer de forma general, als especialistes implicats en el diagnòstic de les malalties infeccioses, les possibilitats de l’EM MALDI-TOF7. S’hi estudien més de 1600 aïllaments que inclouen bacteris grampositius i gramnegatius, aerobis i anaerobis, obtenint un 95,4% d’identificacions correctes. Ja en aquest article s’apunta una qüestió que després s’ha demostrat transcendent per a la utilitat pràctica d’aquesta metodologia: la disponibilitat de bases de dades de microorganismes prou àmplies, tant des del punt de vista qualitatiu (nombre de gèneres i espècies inclosos) com quantitatiu (els autors demostren que la probabilitat d’identificació correcta és major per a aquells microorganismes dels que hi hagi a l’almenys deu perfils diferents introduïts a la base de dades).
Des d’aquest moment, es produeix una ràpida expansió de l’ús d’aquesta tecnologia en Microbiologia Clínica. La primera publicació a Espanya té lloc en 2010, mostrant una correlació amb la metodologia convencional, a nivell d’espècie, de el 100% en grampositius i de el 87,7% al gramnegativos8. Des de llavors, el nombre de publicacions relatives a diferents aspectes de la utilització clínica de l’EM MALDI-TOF, però sobretot a la identificació de microorganismes, ha estat exponencial7-12.
Identificació de bacteris gramnegatius mitjançant MALDI-TOF
els estudis demostren que l’eficàcia en la identificació d’enterobacteris i altres gramnegatius és excel·lent, incloent tant els gramnegatius més habituals en clínica com altres menys freqüents o més complicats d’identificar amb la metodologia clàssica (diferents espècies de Yersinia, Aeromonas, Plesiomonas, Brucella, Francisella, Achromobacter, Stenotrophomonas, Burkholderia …). Com en altres casos, els problemes d’identificació gairebé sempre han estat lligats a insuficiències de les bases de dades més que a limitacions de l’mètode, com ha passat en alguns estudis amb gèneres com Ralstonia, Elizabethkingia o Sphyngobacterium13. Inicialment es van plantejar algunes limitacions que podien tenir més transcendència clínica, com la incapacitat per diferenciar entre Escherichia coli i Shigella14 o la dificultat per diferenciar serovars de Salmonella enterica. Estudis més recents suggereixen que és possible salvar aquestes limitacions. Utilitzant programes informàtics com FlexAnalysis i ClinProTools (Bruker Daltonics GmbH, Alemanya), s’han identificat pics específics que permeten diferenciar E. coli i Shigella en el 90% dels casos15. Altres autors han demostrat que aquesta diferenciació és possible fins i tot sense utilitzar programari addicional, simplement incrementant el nombre i varietat de perfils d’E coli i de Shigella presents a la base de dades de referencia16. Amb aquest mètode, els autors identifiquen correctament 60/64 aïllaments d ‘E. coli i 110/116 aïllaments de Shigella.
A tots els que hem utilitzat l’EM MALDI-TOF per a identificació ens consta que, amb la metodologia habitual, la identificació de Salmonella a nivell de gènere és fiable, però més enllà de l’nivell de gènere ho és molt menys. No obstant això, existeixen ja des de 2004 publicacions que suggereixen l’existència de pics específics que podrien permetre identificar serovars amb més fiabilidad17. Un estudi recent suggereix l’existència de pics que permeten identificar amb fiabilitat el serovar Typhi18. Ja que semblen existir elements diferencials, una ampliació de la base de dades de referència amb un major nombre d’espectres de, al menys, els serovars més freqüents, probablement milloraria la resolució en aquest cas concret.
En altres casos, com és la diferenciació entre espècies de el complex Enterobacter cloacae, la capacitat de l’EM MALDI-TOF està al voltant de l’80%, que sense ser òptima, suposa una millora sensible respecte a la metodologia convencional19.
d’altra banda, com ja s’ha esmentat, la identificació correcta de microorganismes prèviament identificats de manera incorrecta, com el gènere Raoultella, freqüentment identificat pels mètodes clàssics com Klebsiella o com Enterobacter, està permetent una visió més real del seu paper com a patògens, prèviament infravalorado3.
identificació de bacteris grampositius mitjançant MALDI-TOF
La identificació de microorganismes grampositius sol mostrar unes xifres una mica inferiors, a causa en alguns casos a la dificultat per lisar la paret, i en altres de la similitud entre espècies. En general, el mètode directe (aplicació de la colònia juntament amb la matriu sobre la placa de espectròmetre) sol ser suficient per obtenir una bona identificació a partir de colònia, però a vegades pot ser necessària l’extracció prèvia amb etanol / àcid fòrmic per assolir valors d’identificació òptims. En conjunt, ofereix uns resultats excel·lents en la identificació de Staphylococcus aureus i d’altres espècies i subespècies d’estafilococs tant productors com no productors de coagulasa, així com d’enterococs i d’altres patògens menys freqüents com Micrococcus, Gemella, Rothia, etc. Ha demostrat també la seva utilitat en la identificació de bacils grampositius, incloent alguns d’identificació complexa per mètodes clàssics (Listeria, Lactobacillus, Corynebacterium, Nocardia, Actinotignum …) 20.
A més, com en el cas dels gramnegatius , l’ús sistemàtic d’aquesta tecnologia està canviant la consideració com patògens d’alguns microorganismes, com Staphylococcus lugdunensis21. En conjunt, les dades disponibles fan recomanable, en el cas d’estafilococs utilitzar, com en qualsevol altre cas, les bases de dades que ofereixin un major nombre d’espècies i un nombre raonablement alt de perfils per espècie, i utilitzar un mètode d’extracció en lloc de l’aplicació directa, així com aplicar el microorganisme a la placa per duplicat. La reducció de l’score requerit per validar una identificació a nivell d’espècie a valors > 1,7, en lloc de > 2,0 com és habitual, tal com proposen alguns autores22, pot incrementar el nombre d’identificacions a nivell d’espècie, però a canvi d’un descens en l’especificitat la pertinència pot ser discutible.
Probablement el grup dels estreptococs és el que presenta més problemes entre els grampositius. Mentre algunes de les principals espècies patògenes (Streptococcus pyogenes, Streptococcus agalactiae) s’identifiquen amb una alta fiabilitat, Streptococcus pneumoniae (S. pneumoniae) sempre ha estat una font de problemes, per la seva confusió amb altres espècies de el grup mitis. Els principals equips en ús a casa nostra identifiquen S. pneumoniae amb fiabilitat. L’error més habitual és la identificació errònia d’altres estreptococs de el grup mitis com S. pneumoniae, sobretot amb la base de dades de Bruker23. S’han descrit alguns pics com específics de S. pneumoniae, pel que alguns autors recomanen la seva utilització per a corroborar aquestes identificaciones23. No obstant això, l’última biblioteca de referència de Bruker Daltonics (MBT 6903 MSP Library) segueix recomanant la utilització d’altres test (optoquina, solubilitat en bilis) per diferenciar-los.
Fins i tot dins de el mateix grup, la qualitat de la identificació de les diferents espècies pot variar considerablement, com passa dins el grup Streptococcus anginosus.
En tot cas, com passa amb els estafilococs, l’ús d’una base de dades tan àmplia i completa com sigui possible, i la identificació per duplicat i amb protocols que incloguin extracció són recomanables per optimitzar els resultats (fig. 1).
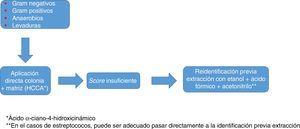 a
a Processament de grampositius, gramnegatius, anaerobis i llevats mitjançant MALDI-TOF.
Identificació de bacteris anaeròbies mitjançant MALDI-TOF
La identificació de bacteris anaerobis, tant grampositius com gramnegatius, té en conjunt un alt grau de fiabilitat, identificant correctament les espècies més habituals (Bacteroides, Prevotella, Porphyromonas, Fusobacterium, Clostridium, Actinomyces …). L’aplicació de l’EM MALDI-TOF en aquest camp ha demostrat, a més, que la identificació bioquímica era molt menys fiable del que es pensaba3. La combinació de senzillesa, rapidesa i fiabilitat converteixen en aquest moment a l’EM MALDI-TOF en el mètode d’elecció per a la identificació rutinària de bacteris anaeròbies en clínica. L’extracció o no de la mostra no sembla modificar sensiblement els resultats, com tampoc ho fa el mitjà de cultiu d’origen. Un factor decisiu, com en altres casos, és l’amplitud de les bases de dades. Ja un dels primers estudis realitzats a Espanya3 mostrava que la major part dels errors en la identificació de bacteris anaeròbies es devien a l’absència de perfils de referència de l’espècie corresponent a la base de dades. Els estudis més antics ofereixen una eficàcia en la identificació al voltant de l’75-80%, mentre estudis més recents, amb bases de dades més completes o amb bases de dades específiques ofereixen xifres superiors a l’90%. La capacitat per identificar ribotipos de Clostridium difficile, suggerida per alguns autors, no ha quedat definitivament demostrada.
Identificació de llevats i fongs filamentosos mitjançant MALDI-TOF
La identificació de llevats ha mostrat des del principi resultats excel·lents, diferenciant fins i tot espècies complicades de discriminar per mètodes convencionals, com és el complex Candida parapsilosis. No obstant això, els resultats en relació amb els fongs filamentosos han estat molt més erràtics. Alguns estudis inicials mostraven bons resultats estudiant selectivament espores, però aquest mètode és poc pràctic en un laboratori clínic, de manera que la majoria dels estudis s’han dirigit a l’estudi conjunt d’espores i hifes. En el cas dels fongs, l’extracció convencional amb àcid fòrmic i acetonitril si millora sensiblement els resultats. Altres mètodes que s’han provat, com la utilització de perles de vidre per a fragmentar les parets, no semblen oferir resultats significativament superiors a l’extracció convencional.
Un problema que plantegen els fongs filamentosos és l’existència de diferències significatives en els perfils proteics obtinguts en funció de l’antiguitat dels cultius, i fins i tot entre diferents subcultius de la mateixa cepa24. La solució a això passa per l’elaboració de bases de dades més àmplies i complexes, que incorporin perfils d’un major nombre de ceps i de cultius de diferent antiguitat. Alguns fabricants recomanen l’extracció després d’un cultiu d’un dia en brou, encara que això repercuteix en un retard de 24 ha l’emissió de l’resultat. Un estudi realitzat a 201.125 demostra que una base de dades ben elaborada i prou complexa és fonamental, i que pot millorar la identificació dels fongs filamentosos fins a xifres similars a les obtingudes amb bacteris i llevats. Desafortunadament, l’elaboració i la validació d’aquestes bases de dades són complexes i no estan a l’abast de molts usuaris, i poques de les desenvolupades estan disponibles. Per això, i també a efectes d’estandardització, és probablement més adequat l’ús de les bases de dades dels fabricants, que han de ser convenientment ampliades.
Actualment Bruker Daltonics, la base de dades de fongs filamentosos, en la seva versió MBT 6903 MSP Library, inclou 25 gèneres i 42 espècies, recomana el cultiu en medi líquid durant una nit, seguida de centrifugació, rentat i extracció amb etanol, àcid fòrmic i acetonitril (fig. 2). MS-VITEK, en la seva versió 3.0, inclou 32 gèneres i 81 espècies, i SARAMIS en la seva versió RUO 4.13, inclou una base de dades més àmplia, amb 45 gèneres i 168 espècies. bioMérieux no recomana el cultiu en medi líquid, sinó una extracció convencional amb etanol, àcid fòrmic i acetonitril (fig. 2). Malgrat tot, un estudi recent usant la metodologia recomanada per Bruker identifica correctament, a nivell d’espècie, només el 72% dels aislados26, i un estudi molt recent amb VITEK 3.0 identifica correctament el 66,8% de 318 aïllats, a causa sobretot a mancances de la base de datos27. Per tant ara per ara, la identificació de fongs filamentosos mitjançant EM MALDI-TOF no pot substituir encara completament a la metodologia d’identificació convencional.
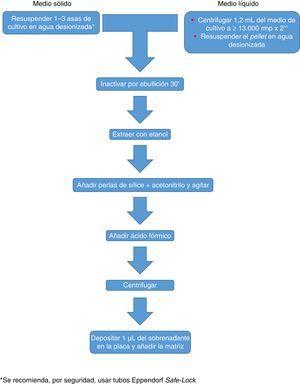 a
a Processament de micobacteris recomanat per al sistema MALDI Biotyper.
identificació de micobacteris mitjançant MALDI-TOF
Les limitacions de la metodologia tradicional per a la identificació de micobacteris, sobre tot pel que fa a el temps de resposta, han fet que la pràctica totalitat dels laboratoris s’hagin decantat pels mètodes moleculars d’identificació. La utilització de l’EM MALDI-TOF per a la identificació de micobacteris suposa una alternativa igualment ràpida i més barata que les tècniques moleculars, però mostra peculiaritats en diversos aspectes. D’una banda, els mètodes directes d’anàlisi, o que impliquin una alta concentració o manipulació dels microorganismes prèviament a la seva inactivació, no són recomanables per una qüestió de seguretat biològica. La utilització d’un mètode d’extracció que garanteixi el trencament de les cèl·lules millora la qualitat dels espectres, i incrementa la seguretat de l’procediment.
Diversos factors poden influir en l’eficàcia de la identificació de micobacteris mitjançant EM MALDI -TOF. Com també s’ha demostrat amb els fongs, micobacteris poden presentar perfils proteics molt diferents en funció de l’antiguitat dels cultius, per la qual cosa és important per al diagnòstic incloure en les bases de dades perfils proteics de cultius de diferent antiguitat per a cada microorganisme . S’ha parlat també de la possible formació de polímers que emmascaren la mida real de les proteïnes característiques utilitzades per a la identificació. Tot això fa de la identificació de micobacteris un procediment menys previsible pel que fa als seus resultats que la d’altres bacteris, com demostra l’heterogeneïtat de les dades publicades. En les últimes actualitzacions, les biblioteques han experimentat importants millores. Així, per exemple, la biblioteca de micobacteris 3.0 de Biotyper inclou ja 149 espècies, i la versió 3.0 de VITEK MS incorpora 48 noves espècies pel que fa a la versió anterior.
El procediment inicialment recomanat per Bruker incloïa una sèrie de passos (s’ajusti a una determinada densitat òptica, diversos passos de centrifugació i resuspensió) que suposaven un risc biològic probablement innecessari, pel que han estat substituïts per un sol pas de tractament amb calor. La manera més recomanable actualment inclou diversos passos d’escalfament i rentat, seguits d’un tractament amb boles de sílice i acetonitril. Posteriorment són tractades amb àcid fòrmic, aplicades a la placa i cobertes amb la matriu (fig. 3). Per contra, el mètode propugnat per bioMérieux part d’un tractament inicial amb etanol a el 70% i boles de sílice, després del qual la mostra és extreta amb àcid fòrmic i acetonitril, aplicada a la placa i coberta amb la matriu (fig. 4 ).
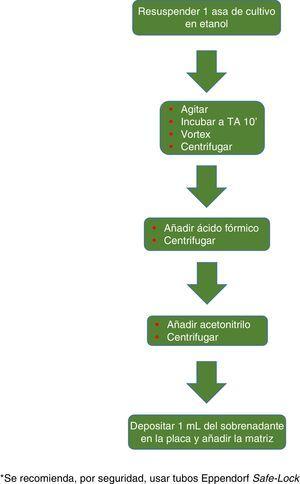 a
a Processament de micobacteris recomanat per al sistema VITEK MS.
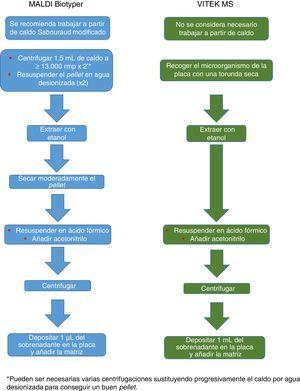 a
a Processament de fongs recomanat per als sistemes MALDI Biotyper i VITEK MS .
Un estudi comparatiu entre els dos mètodes dut a terme recienteme nte28 mostra que encara que la identificació tendeix a ser una mica millor partint de mig 7H10 que de Lowenstein Jensen, les diferències són petites en ambdós sistemes. No obstant això, demostra que el mètode de processament de la mostra és crític, i no és intercanviable entre sistemes. Les mostres obtingudes amb el mètode d’extracció de Bruker donen millors resultats en el seu espectròmetre i les processades amb el mètode de Vitek ho fan tant amb SARAMIS com en el propi Vitek MS, tot i que la diferència en aquest cas és menor. En conjunt, Vitek MS sembla veure menys afectat pel mètode d’extracció. Pel que fa a l’eficàcia en la identificació, Bruker Biotyper va identificar amb un score ≥1,8 un 81,5% dels aïllats, mentre SARAMIS i Vitek MS 3.0 van identificar amb una fiabilitat ≥90% 1 85,4 i un 89,2 % respectivament. Utilitzant aquests mateixos criteris, un 22,6% dels aïllats que van ser identificats per Bruker Biotyper van necessitar ser extrets en més d’una ocasió per aconseguir la identificació, el que va passar en un 21,5% amb SARAMIS i en un 14,2% amb Vitek MS 3.0.
En relació amb altres microorganismes amb estructura parietal propera a la dels micobacteris, durant temps s’ha considerat que la seva identificació era problemàtica a causa de la composició de la seva paret. No obstant això, s’ha demostrat que, si es disposa d’una base de dades amb suficients referències, un mètode d’extracció similar a l’usat amb micobacteris pot oferir bons resultados29.
Totes les dades referides anteriorment suggereixen que l’espectre de microorganismes susceptibles d’identificació mitjançant EM MALDI-TOF és extraordinàriament ampli i que, amb comptades excepcions, el factor limitant de la capacitat de l’EM MALDI-TOF s’assenta gairebé sempre més en la completesa de les bases de dades de referència que en la capacitat de el mètode per obtenir perfils fiables de gairebé qualsevol microorganisme bacterià susceptible de créixer en mitjans de cultiu. De fet, les empreses fabricants són conscients de el problema; així, l’última biblioteca de referència publicada per Bruker Daltonics (MBT 6903 MSP Library) incorpora 938 nous perfils, dels quals un 17,2% van orientats a cobrir nous gèneres i espècies (15 nous gèneres i 96 noves espècies en concret), especialment anaerobis i fongs, però un 82,8% van orientats a incrementar la diversitat de perfils dins d’espècies ja contemplades en biblioteques prèvies. De la mateixa manera, la versió 3.0 de VITEK MS inclou 242 noves espècies bacterianes, entre les quals es troben 48 noves de micobacteris i 15 de Nocardia, i 55 noves de fongs, amb una mitjana global de més de 10 aïllats i 25 espectres per espècie .
identificació de microorganismes, per MALDI-TOF, a partir de mostra directa
La possibilitat d’identificació de microorganismes prèviament a el creixement de colònies ha estat, des del principi, un objectiu molt atractiu, pel que suposa a l’hora de poder instaurar un tractament empíric més orientat, reduir l’emergència de resistències, i optimitzar el gasto30. L’EM MALDI TOF, aplicada a mostra directa, s’ha convertit en una eina de gran utilitat en infeccions greus amb alta taxa de morbimortalitat com bacterièmies i fungemias, si bé el treball directe sobre mostres presenta inconvenients que poden limitar la utilitat de la tècnica. En mostres amb baixa densitat microbiana no és possible realitzar una correcta identificació, ja que la quantitat de proteïnes bacterianes presents és insuficient. Una cosa semblant passa amb les mostres polimicrobianes, ja que es pot generar un espectre proteic aberrant, com a resultat de la barreja de diversos perfils, o directament ignorar-se el microorganisme que estigui en una proporció menor. S’han desenvolupat algunes millores de programari que de vegades poden detectar, i fins i tot identificar per separat els microorganismes implicats, però requereix ser acuradament estudiat i probablement depurat per tenir utilitat real. En la major part dels mètodes que treballen directament sobre mostra, i davant la possible presència de microorganismes intracel·lulars, és recomanable usar mètodes que incloguin lisi cel·lular.
No totes les mostres són vàlides per a aquest tipus d’estudi. La mostra ideal és aquella que procedeixi d’una àrea habitualment estèril, que pugui albergar altes concentracions de microorganisme i en la qual no hi hagi limitacions significatives pel que fa a l’volum de mostra. Les mostres procedents d’àrees habitualment colonitzades (pell, femta, aparell respiratori alt …) generaran amb tota probabilitat perfils aberrants. Una qüestió tècnica a tenir en compte quan es treballa directament sobre mostra és el volum de mostra disponible, que en casos com orina o hemocultius no sol ser un problema, però en altres amb líquid cefaloraquidi sí que pot ser-ho, i que serà tant més important com menor sigui la concentració bacteriana. A més, mostres amb alt contingut proteic d’origen aliè a l’microorganisme també poden generar problemes a l’hora de la seva interpretación31.
No obstant això, l’avantatge que suposa, especialment en els hemocultius, avançar 24 h la identificació de l’ microorganisme i orientar de manera més específica el tractament, ha fet que l’ús directe de l’EM sobre mostra, especialment en aquests casos, s’hagi generalitzat.
Identificació directa a partir de mostres d’orina
l’orina és una de les mostres que millor s’ajusten a les condicions ideals per treballar amb EM MALDI-TOF sobre mostra directa. És un líquid estèril en condicions fisiològiques, la càrrega bacteriana en l’orina infectada és alta en la majoria dels casos, i les limitacions pel que fa a l’volum de mostra ia l’obtenció de noves mostres si cal, són mínimes.
s’han publicat estudis amb resultats excel·lents, amb concordances en la identificació que arriben a l’90-95% 32,33 en comparació amb els sistemes automatitzats.S’han descrit diferents protocols de processament de mostres, coincidint tots ells en la necessitat d’un cribratge previ (citometria de flux, tinció de Gram) per discriminar entre mostres positives i negatives, i limitar l’ús de l’EM a les mostres inicialment positivas34.
amb petites variacions, la preparació de les mostres sol consistir en sotmetre-les a una sèrie de centrifugacions i rentats amb aigua desionitzada, portant a terme posteriorment el procediment convencional de l’EM MALDI-TOF. S’han realitzat modificacions incorporant a l’alíquota de la mostra SDS a l’10% 33 per millorar l’alliberament de les proteïnes, o Tween-8034 per millorar els resultats. Els resultats obtinguts solen ser millors amb gramnegatius que amb grampositius i fongs, millorant molt la fiabilitat quan els recomptes són elevats, per sobre de 105UFC / ml.
Un inconvenient d’aquest mètode ha vingut sent la necessitat de dur a terme l’estudi de sensibilitat mitjançant la metodologia convencional. Un estudi recent suggereix que, un cop obtinguda la identificació per EM MALDI-TOF, el mateix sediment es pot usar per realitzar un antibiograma disc placa amb excel·lents resultats, el que permet reduir en 24 h l’estudi completo35. L’important canvi organitzatiu que la introducció d’aquesta metodologia suposa, i el que es tracti d’infeccions en què, sovint, avançar unes hores el diagnòstic no és crític per al maneig de l’pacient, han fet que el diagnòstic de la infecció urinària mitjançant EM MALDI-TOF no hagi tingut la penetració que ha tingut, per exemple, en el cas dels hemocultius. Cal aconseguir tant una major sensibilitat com una major normalització dels mètodes per plantejar la seva introducció en la rutina clínica.
Identificació directa a partir d’hemocultius
La instauració d’un tractament empíric correcte és decisiva en l’evolució de les bacterièmies. La probabilitat que el tractament instaurat sigui correcte, serà més gran com més exacta i concreta sigui la informació que siguem capaços de proporcionar amb rapidesa. En aquest aspecte, la possibilitat oferta per l’EM MALDI-TOF d’obtenir una identificació fiable en un curt període de temps després de la positivització de l’hemocultiu suposa un avanç evident.
S’han descrit nombrosos protocols de processament de l’hemocultiu (centrifugació diferencial, lisi cel·lular i extracció de proteïnes per mètodes químics, separació mitjançant gel …). La majoria d’ells estan basats en la lisi i eliminació dels components cel·lulars, bé mitjançant centrifugacions i rentats, en els quals s’utilitzen diferents compostos com saponina, clorur d’amoni o SDS, o bé mitjançant l’ús de tubs amb gel separador de sèrum i activador de la coagulació. En definitiva, el que es busca amb aquests mètodes és aïllar i concentrar els microorganismes fins a arribar a el menys 105-107UFC / ml, concentració a la qual la quantitat de proteïnes és suficient per generar perfils adequats a l’EM MALDI-TOF.
en el cas dels hemocultius ha un procediment comercial homologat (Sepsityper, Bruker Daltoniks GmbH, Alemanya), consistent en l’addició d’una solució de lisi a una alíquota de 1ml de l’hemocultiu, després d’això la mostra és sotmesa a diversos passos de rentat i centrifugació i, finalment, a una extracció convencional amb etanol i àcid fórmico36.
Els resultats de l’processament directe d’hemocultius són en general bons, amb percentatges d’identificació correcta de l’80-90%, tot i que amb alguns matisos. La identificació dels gramnegatius sol ser correcta en el 90-95% dels casos, mentre en grampositius és molt més heterogènia, oscil·lant entre xifres similars a les de gramnegatius i xifres al voltant de l’50%. Dins d’aquests, plantegen problemes de fiabilitat sobretot els estreptococs de el grup viridans i els estafilococs no productors de coagulasa.
Els resultats en casos d’Fungemia, en els primers estudis, eren decebedors. Una correcta extracció, indispensable en aquest cas, porta a xifres d’identificació superiors a l’90%, homologables amb les obtingudes en bacteriemias37. S’ha de tenir en compte la possible repercussió de determinats components d’alguns hemocultius, com el carbó actiu, que pot interferir la identificació. Així mateix, un temps d’incubació prolongat de l’hemocultiu pot repercutir negativament en els espectres obtinguts. No obstant això, ja que el procediment habitual és realitzar l’EM en, com a màxim, unes hores a partir de la positivització, i més de l’80% dels hemocultius positius significatius ho són en les primeres 48 h, l’impacte clínic d’aquest aspecte no sembla transcendent.
A l’hora de valorar l’heterogeneïtat dels percentatges d’identificació correcta en els hemocultius, ha de tenir-se en compte que no hi ha un criteri homogeni respecte a com valorar els scores obtinguts. Alguns autors consideren una identificació correcta amb valors > 1,7. Altres disminueixen aquesta exigència fins a 1,5, però inclouen requisits com que la identificació es repeteixi en les dues o tres primeres posicions de la llista d’identificacions possibles, o que entre les dues primeres opcions que ofereix l’EM MALDI-TOF hi hagi una diferència de score de al menys 0,3 punts.
en general, no som partidaris de reduir el nivell d’exigència en la identificació correcta en els hemocultius, ja que l’existència d’un major percentatge de “no identificacions” suposarà sempre un menor risc, des del punt de vista clínic, que la proliferació d’identificacions incorrectes, que podrien condicionar tractaments inadequats.
quant a la utilització de mètodes comercials de processament, com Sepsityper, els resultats en general són similars en relació amb els mètodes de processament manuals. Cada laboratori ha de prioritzar entre la major normalització i l’estalvi en temps de processament que propicien aquests mètodes, o l’estalvi econòmic (al voltant de 1 € / mostra) que suposa la utilització d’un mètode manual.
En conjunt, l’EM MALDI-TOF és un mètode ràpid i fiable per a la identificació directa de microorganismes en hemocultius. La seva combinació amb mètodes que permeten detectar mecanismes de resistència a determinats antimicrobians, permet oferir una informació clínica valuosa en un temps sensiblement inferior (24-48 h menys) a l’requerit per la metodologia convencional. Probablement el principal punt a dilucidar, en aquest moment, és la forma d’integrar aquesta nova activitat en els laboratoris de Microbiologia, de manera que s’optimitzi la informació oferta sense suposar una sobrecàrrega excessiva en serveis, en general, no precisament sobrats de personal tècnic i amb estructures horàries molt diverses.
Identificació directa a partir d’altres mostres
El nombre d’estudis relatius a l’aplicació directa de l’EM MALDI-TOF a altres mostres biològiques és més reduït. Tal com es comenta en un altre apartat, la utilitat de l’EM MALDI-TOF en aquestes mostres ve determinada per diversos factors: d’una banda, la mostra ha de procedir d’una àrea estèril en condicions fisiològiques, i en la qual la infecció, quan aparegui, tendeixi a ser habitualment monomicrobiana. D’altra banda, són crucials tant la càrrega bacteriana present a la mostra, com el volum de mostra disponible per a l’estudi. Això fa que moltes mostres diferents de l’orina i el hemocultiu plantegin problemes, pel baix volum habitualment disponible (LCR, exsudats purulents), per la baixa concentració de microorganismes (LCR, líquid peritoneal en pacients sotmesos a diàlisi peritoneal) o per la presència de quantitats importants de proteïnes no bacterianes que poden alterar els perfils o interferir en la ionització de les proteïnes bacterianes (exsudats purulents).
la baixa sensibilitat en aquells productes en què la càrrega bacteriana no és molt alta, i el volum de mostra disponible, sovint limitat, fan que, en general, siguin preferibles per al diagnòstic ràpid les tècniques moleculars, tot i la limitació que el ventall de microorganismes detectables en un sol test és molt menor que amb l’EM MALDI -TOF. S’han comunicat ocasionalment diagnòstics etiològics de meningitis bacterianes per EM MALDI-TOF directa sobre LCR, però un estudi comunicat a l’21erECCMID indicava que, sobre 183 mostres de LCR, de les quals 14 van ser positives pel mètode convencional, cap va ser positiva per EM MALDI -TOF directa sobre muestra38, el que reafirma el fet que, des del punt de vista de la sensibilitat, l’EM MALDI-TOF no pot competir amb la tècniques moleculars. En altres tipus de mostra l’experiència és molt limitada, no existeixen protocols publicats ni dades respecte a sensibilitat i especificitat, i per tant la utilització clínica de l’EM MALDI-TOF, en aquest moment, no és pertinent.
Mitjans cromogénics
Des de l’aparició del primer mig cromogènic (MC), fa més de 30 anys, s’han convertit en una eina molt útil per a l’aïllament diferencial de microorganismes patògens. Existeixen actualment multitud de MC comercialitzats per a la identificació de bacteris i fongs, així com per a l’estudi d’alguns mecanismes de resistència a antimicrobians.
El fonament d’aquests mitjans en la inclusió d’un substrat cromogènic que, a l’ésser hidrolitzat per un enzim específic present al microorganisme, dóna lloc a una colònia de coloració característica, permet la seva diferenciació.Sovint són a més mitjans selectius, que inhibeixen en major o menor mesura el creixement d’altres microorganismes, afavorint la detecció de les colònies acolorides. Els principals substrats dels MC comercials són derivats indólicos39 que poden ser hidrolitzats per galactosidasas o glucosidases, produint-derivats poc tòxics i que no inhibeixen el creixement bacterià.
Detecció de microorganismes grampositius
S’han dedicat grans esforços a el desenvolupament de mètodes per a la detecció de S. aureus resistent a meticil·lina (MRSA), donada la seva importància en infecció nosocomial. En aquest context, l’ús de MC ha cobrat gran importància per al ràpid aïllament i identificació d’aquest bacteri. Existeixen en el mercat nombrosos MC per a la detecció de MRSA. Un estudi reciente40 compara tres d’ells: chromID MRSA SMART (SMART), chromID MRSA fi rst generation (chromID) i Brilliance MRSA (OX2) per al cribratge de 1.220 mostres de pacients hospitalitzats. La detecció en aquests mitjans es va corroborar mitjançant EM MALDI-TOF, difusió en agar amb discos de cefoxitina i PCR comercial per Meca i Mecc. La sensibilitat a les 24 h va ser millor per al medi SMART pel que fa a el medi chromID, però no hi va haver diferències significatives amb el medi OX2. No obstant això, els autors indiquen que encara cal, per obtenir uns resultats òptims, l’enriquiment en brou durant 24 h abans de la inoculació en qualsevol dels mitjans estudiats.
Xu et al.41 fan una revisió de 5 dels mitjans més usats per a la detecció de MRSA. El que millors resultats ofereix en aquest estudi és el Brilliance MRSA 2 (Oxoid Ltd, Termofisher, EE. UU.), Amb una sensibilitat de el 65,7% i una especificitat de el 99,8%. El rendiment d’aquest mitjà millora si s’inocula la mostra en un brou d’enriquiment abans de sembrar-la en el MC, amb una sensibilitat de 100% i una especificitat de el 99,1%. El medi CHROMagar MRSA, mostra una sensibilitat de l’95% després de 24 h d’incubació, que arriba a un 100% si s’incuba 48 h. L’especificitat en tots dos casos és de 100%. El medi BBL Chromo agar MRSA inclou cefoxitina en la seva composició, el que simplifica la interpretació dels resultats. Aquest mitjà va ser estudiat utilitzant aïllaments de MRSA obtinguts de hemocultius, amb una sensibilitat i especificitat de l’97,6 i el 99% respectivament. MRSA Select ser avaluat utilitzant 652 aïllats procedents de hemocultius després 18-24 h d’incubació a 35 ° C, amb una sensibilitat de el 99% i una especificitat de l’98%, que augmentava a el 99% quan es combinava amb la prova de la coagulasa. El medi chromID MRSA, que també incorpora cefoxitina en la seva composició, va ser estudiat en aïllaments procedents de ferides i de hemocultius. En hemocultius mostra una sensibilitat de el 97,8%, a les 24h, que augmenta a un 100% a les 48 h, i una especificitat de l’99,7% que es manté (99,6%) a les 48 h. En ferides, la sensibilitat i especificitat van ser de l’88,9 i de el 100% a les 24 hi de el 100% en els dos casos a les 48 h.
L’establiment de comparacions entre aquest tipus de mitjans de cultiu és complex, ja que hi ha molts factors, a part de el propi medi de cultiu, que poden influir en els resultats (tipus de mostra, concentració de l’inòcul, temps d’incubació …). No obstant això, les dades de sensibilitat i especificitat disponibles suggereixen que qualsevol dels mitjans abans esmentats pot ser utilitzat de rutina per a la detecció de MRSA amb alta fiabilitat.
Un altre grup de bacteris que ha adquirit importància en la infecció nosocomial són els enterococs resistents a vancomicina (VRE), fins al punt que els CDC recomanen l’estudi de portadors en els centres amb alta prevalença. L’estudi de portadors de VRE s’ha dut a terme utilitzant agar bilis esculina amb azida i vancomicina (Beav). El seu ús implicava un retard diagnòstic de 48 h, a més de la necessitat de realitzar proves complementàries per a una correcta identificació. Per agilitzar aquest procés s’han desenvolupat diversos MC. Un estudi recent compara 5 MC per VRE usant 400 mostres de heces42. Els mitjans utilitzats van ser intra Colorex VRE (BioMedDiagnostics, White City, OR), chromID VRE (bioMérieux, Marcyl’Étoile, France), VRESelect (Bio-Rad, Marnes-la-Coquette, France), HardyCHROM VRE (Hardy Diagnostics, Santa Maria, CA) i Spectra VRE (Remel, Lenexa, KS), utilitzant agar Beav i brou Beav com a mètode de referència. La lectura es va realitzar a les 24 h. Els resultats dels cinc mitjans mostren una sensibilitat d’entre el 89,9 i el 94,9%, més alta en tots els casos que la mostrada per l’agar Beav (84,9%). Els millors dades de sensibilitat i especificitat els segueix proporcionant el brou Beav, però a costa d’un retard diagnòstic de fins a 48 h pel que fa als MC. El MC més sensible en aquest estudi va ser chromID VR (94,9%), tot i que sense diferències estadísticament significatives amb la resta.
L’especificitat i la capacitat per diferenciar espècies d’Enterococcus van ser similars per als cinc MC. El medi intra i Colorex VR va tenir l’especificitat més baixa amb un 98,8% mentre que les de les altres quatre plaques van ser de 99,7%. Les característiques de sensibilitat i especificitat d’aquests mitjans els fa recomanables per a la detecció de portadors de VRE quan aquesta sigui pertinent. Són, si més no, perfectament equiparables a l’agar Beav, disminuint el temps de detecció en 24-48 h.
Detecció de microorganismes gramnegatius
El 1979, Kilian i Blow descriuen un nou mitjà de cultiu que utilitza la β- glucoronidasa com a substrat per a la detecció directa d’E coli en urocultius. Els estudis de rendibilitat indicaven que aquest mètode suposava un estalvi econòmic de l’46% i una reducció de el temps d’identificació d’un 64% respecte als mètodes convencionales39. Des de llavors s’han desenvolupat nombrosos MC amb diferents substrats per a la identificació dels principals uropatògens. Uns dels mitjans comercialitzats recentment és chromID CPS Elite (bioMérieux, Durham, NC) que permet la identificació directa d’E coli, i la identificació presumptiva d’Enterococcus spp., D’algunes Enterobacteriaceae i dels bacteris de el grup Proteae, encara que en aquests casos és necessària la confirmació de la identificació mitjançant proves bioquímiques o EM MALDI-TOF.
s’ha avaluat l’efectivitat d’aquest mitjà per a la detecció de uropatógenos43, valorant el temps requerit per al diagnòstic i els consumibles utilitzats en comparació amb la utilització d’agar sang i agar MacConkey. La concordança d’aquest mitjà amb la metodologia convencional va ser de l’88% per a orines clínicament significatives, de l’el 74% per orines amb creixement no significatiu, el 69% per a les contaminades i un 95% per plaques sense creixement bacterià. Les principals discrepàncies es van deure a el creixement en agar sang, però no en el MC de bacteris grampositius, en general amb poca rellevància clínica en urocultius, com Staphylococcus spp. i Lactobacillus. Pel que fa a el temps transcorregut entre la sembra de la mostra i la identificació de les colònies, la diferència no va ser estadísticament significativa si es comparen els dos mitjans de forma global (27,2 h de mitjana per al mètode convencional enfront de 26,6h per l’MC), però sí que ho va ser en aquelles orines en què va créixer E. coli en cultiu pur (27,1 h al mètode convencional enfront de 24,4 h en chromID, p
0,0001). Els autors addueixen que aquesta millora de temps és deguda a la facilitat i fiabilitat de la identificació pel color de la colònia, que evita la necessitat de mètodes d’identificació complementaris.
Els mitjans chromID ™ CPS (CPS4) (bioMérieux, St . Laurent, QC) i UriSelect ™ abril (URS4) (Bio-Rad, Montreal, QC), han estat evaluados44, sobre 903 mostres d’orina, en comparació també amb el mètode convencional d’agar sang i agar MacConkey. La concordança amb el mètode convencional va ser de l’89,3 i 89,5% per a URS4 i CPS4 respectivament. Si es consideren només aquelles mostres en què el creixement es va considerar clínicament significatiu, aquesta concordança augmentar a un 93% en el cas de URS4 ia un 93,1% per al medi CPS4.
En tots els casos , els autors coincideixen que la major avantatge de l’ús de MC és l’agilització el procés d’identificació, a l’reduir-se la necessitat de proves complementàries, el que permet realitzar un diagnòstic etiològic amb més rapidesa.
Donada la utilitat demostrada d’aquesta eina diagnòstica, diversos grups estan dissenyant nous MC, alguns encara no comercialitzats, que poden suposar una alternativa interessant en el futur. S’ha dissenyat un nou medio45 per a la detecció de Bacteroides, que inclou en la seva formulació 3,4-ciclohexanoesculetina-β-D-glucòsid, que és la diana d’acció de la β-glucosidasa de Bacteroides, i que dóna lloc a l’aparició de colònies de color negre. Aquest mitjà s’ha comparat amb l’agar bilis esculina en 100 mostres fecals. El MC va permetre la identificació de B. fragilis en 34 mostres, mentre que en agar bilis esculina només es va detectar aquest microorganisme en 19 mostres. A més, el MC va resultar molt més selectiu, ja que en cap cas es va observar creixement d’espècies no pertanyents a l’gènere Bacteroides, mentre en agar bilis esculina es va observar creixement de microorganismes pertanyents a altres gèneres (enterobacteris, Clostridium, Candida …) en 34 ocasions . Els autors estudien a més la capacitat d’aquests MC, suplementats amb meropenem i amb metronidazol, per detectar la presència de Bacteroides resistents a un o altre antimicrobià, demostrant que en tots dos casos són capaços de detectar soques resistents amb una sensibilitat de 100% i una especificitat de, al menys, el 85%.
Un altre mitjà innovador desenvolupat és el CHROMOagar Yersinia enterocolitica que presenta com a avantatge enfront de l’mig Cefsulodina-Irgasan-Novobiocina la possibilitat de creixement de soques de Y. enterocolitica serovar O3 i d’I psedotuberculosis, el creixement es pot veure inhibit en el medi CIN46.
Detecció de llevats
Tot i que els primers MC desenvolupats per fongs, a principis dels 90, van tenir poc èxit en la seva aplicació clínica per la seva limitada capacitat de diferenciació, els MC per micologia desenvolupats en els últims 15 anys han tingut un èxit molt més gran, ja que permeten una identificació presumptiva ràpida de diferents espècies de llevats, un millor estudi de cultius mixtos, i la identificació precoç d’espècies associades a resistència a antifúngics. Els mitjans desenvolupats es basen, com en els casos anteriors, en la presència de substrats per a una o diverses enzims (β-N-acetil hexosaminidasa, i en alguns casos, β-glucosidasa o fosfatasa), que permeten la identificació ràpida, com a mínim , de Candida albicans, i en nombrosos casos la identificació definitiva o presumptiva de Candida glabrata, Candida lusitaniae, Candida kefyr, C. parapsilosis, Candida tropicalis i Candida krusei. Els estudis disponibles i la ja prolongada experiència amb aquest tipus de mitjans demostren una alta especificitat d’identificació i una bona sensibilitat. No obstant, donada la variabilitat de color i morfologia que pot aparèixer en aïllaments de la mateixa espècie, la identificació ha de sempre ser considerada presumptiva. Un estudi recent sobre més de 5.600 mostres clíniques positives per llevats, demostra que més de el 8% eren cultius mixtos, la detecció és molt més probable si s’utilitzen MC, i proposa un algoritme que combina MC i EM MALDI-TOF com a procediment més adequat de identificación47.
conflicte d’interessos
Els autors declaren no tenir cap conflicte d’interessos.